シャローショットガンメタゲノムシーケンス

シャローショットガンメタゲノムシーケンス (SSMS) は、ショットガンメタゲノムシーケンスと16S/18S/ITSアンプリコンシーケンスとの間にあるギャップを埋めるものです。シャローショットガンメタゲノムシーケンスでは、微生物の分離や培養の必要性を回避しながら、サンプルに含まれるすべての生物の全ゲノムDNAについて分類学的な情報を提供します。これは、全微生物の99%近くが実験室内で培養できないため、非常に重要なことです。ショットガン・メタゲノミックが高解像度マイクロバイオーム解析のゴールドスタンダードである一方、SSMSはアンプリコンシーケンスに代わる強力な手法です。SSMSは次世代シーケンサー(NGS)を使用し、PCRバイアスがほとんどない、あるいは増幅領域によって制限された、異なる分類群にわたる種レベルの分類学を提供します。
ノボジンのシャローショットガンメタゲノムシーケンスサービスは、3Gb未満の生データでメタゲノムサンプルの多様性を検出、特徴づけ、比較を提供します。このアプローチは、コミュニティ微生物生態学研究、微生物と微生物の相互作用や病原性の理解など、様々な分野に適しています。ノボジーンでは、さまざまな研究目的に適した、論文化可能な結果である高品質データを提供しています。
シャローショットガンメタゲノムシーケンスの応用例
シーケンス
メタゲノミックサンプルから、よりPCRバイアスの少ない、より高度な分類学的プロファイリングを提供することにより、シャローショットガンメタゲノムシーケンスは、様々な研究テーマで使用することができます。
- 微生物の種レベルのアノテーション。
- 微生物-宿主関係を理解するために、常在菌または病原性微生物を検出する。
- ヒトの健康に関連する生理学的な体液や組織からのサンプルを分析する。
- 大規模なヒトマイクロバイオームコホート研究に適しています。
ノボジーンの強み
- シャローショットガンメタゲノムシーケンスでは、単離や培養の必要なく、1回のランで複数の微生物をシーケンスできます。同様に、シャローショットガンメタゲノムシーケンスでは、原核生物または真核生物のいずれかの特定の標的領域の増幅を必要としないため、PCRバイアスが限定的です。シーケンスの結果は微生物の多様性と構成に関する包括的な情報を提供します。
- ノボジーンは、ショートリードシーケンスにより様々なサンプルから情報を得ることができるメタゲノムシーケンスサービスを提供した豊富な経験を有しています。ノボジーンでは、シャローショットガンメタゲノムシーケンスサービスにより、Kraken2によるメタゲノムデータのリードベース解析を通じて、低いシーケンスデプスで実験デザインの柔軟性と費用対効果を実現しています。
サンプル要件
| Sample Type | Amount | Volume | Concentration | Purity |
| Total DNA | ≥ 200 ng | ≥20 μL | ≥ 10 ng/μL | A260/280 = 1.8-2.0 |
注:表示されているサンプル量は参考値です。詳細な情報については、お問い合わせください。
仕様:シーケンスと解析
| Sequencing Platform | Illumina NovaSeq 6000 |
| Read Length | Pair-end 150 bp |
| Recommended Sequencing Depth | 1-3 Gb |
| Standard Data Analysis |
|
注:記載されているデータ量・解析内容は参考値です。詳しくはサービス仕様書をダウンロードしてご確認ください。詳しい内容については、お問い合わせください。.
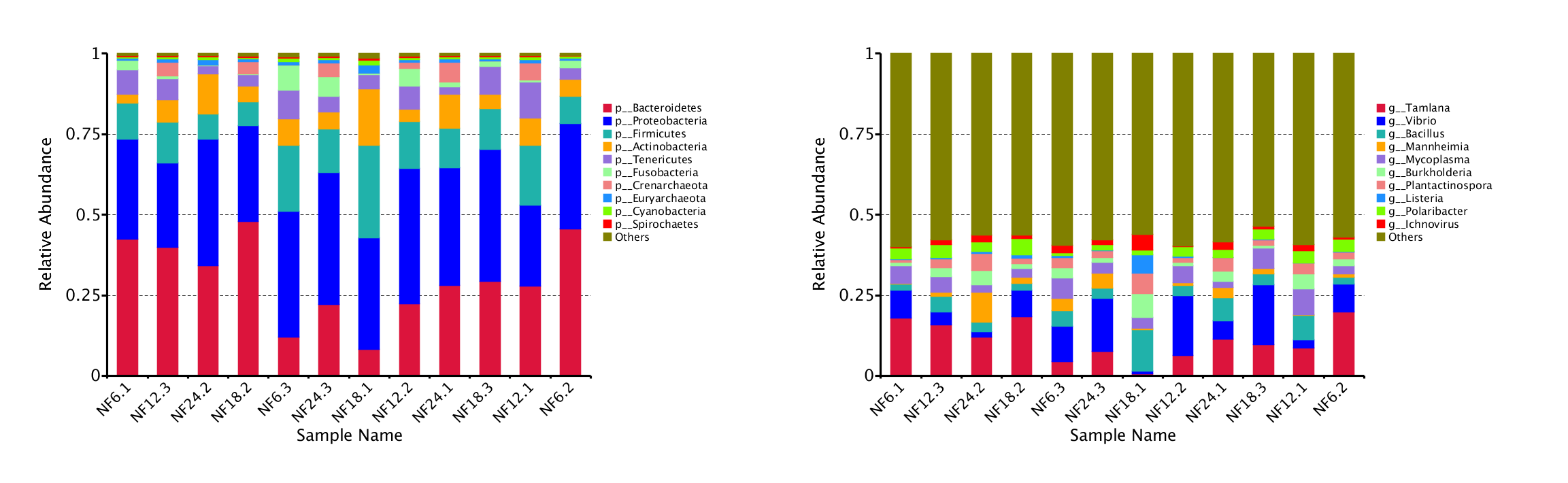
Taxonomic Abundance Bar Graph
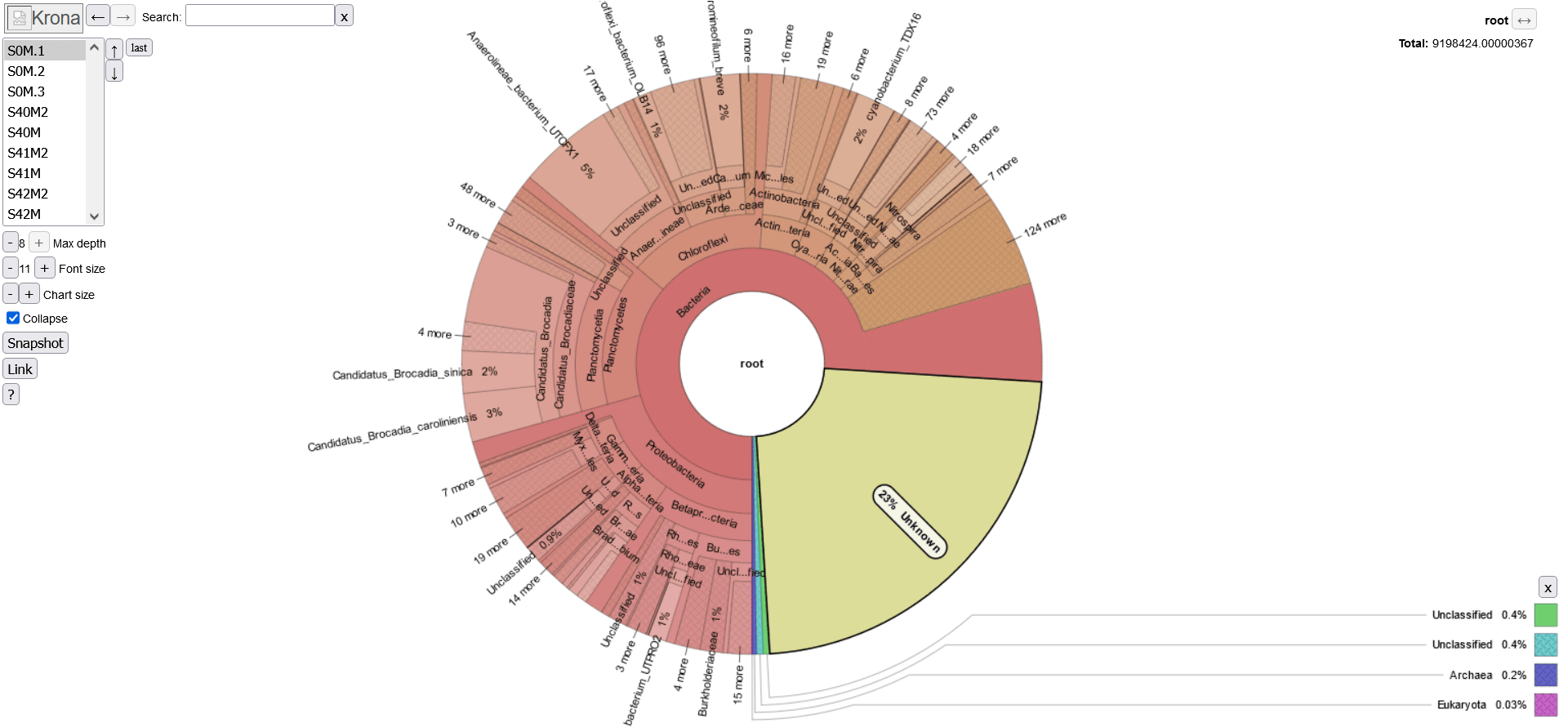
Krona For Microbiome Abundance
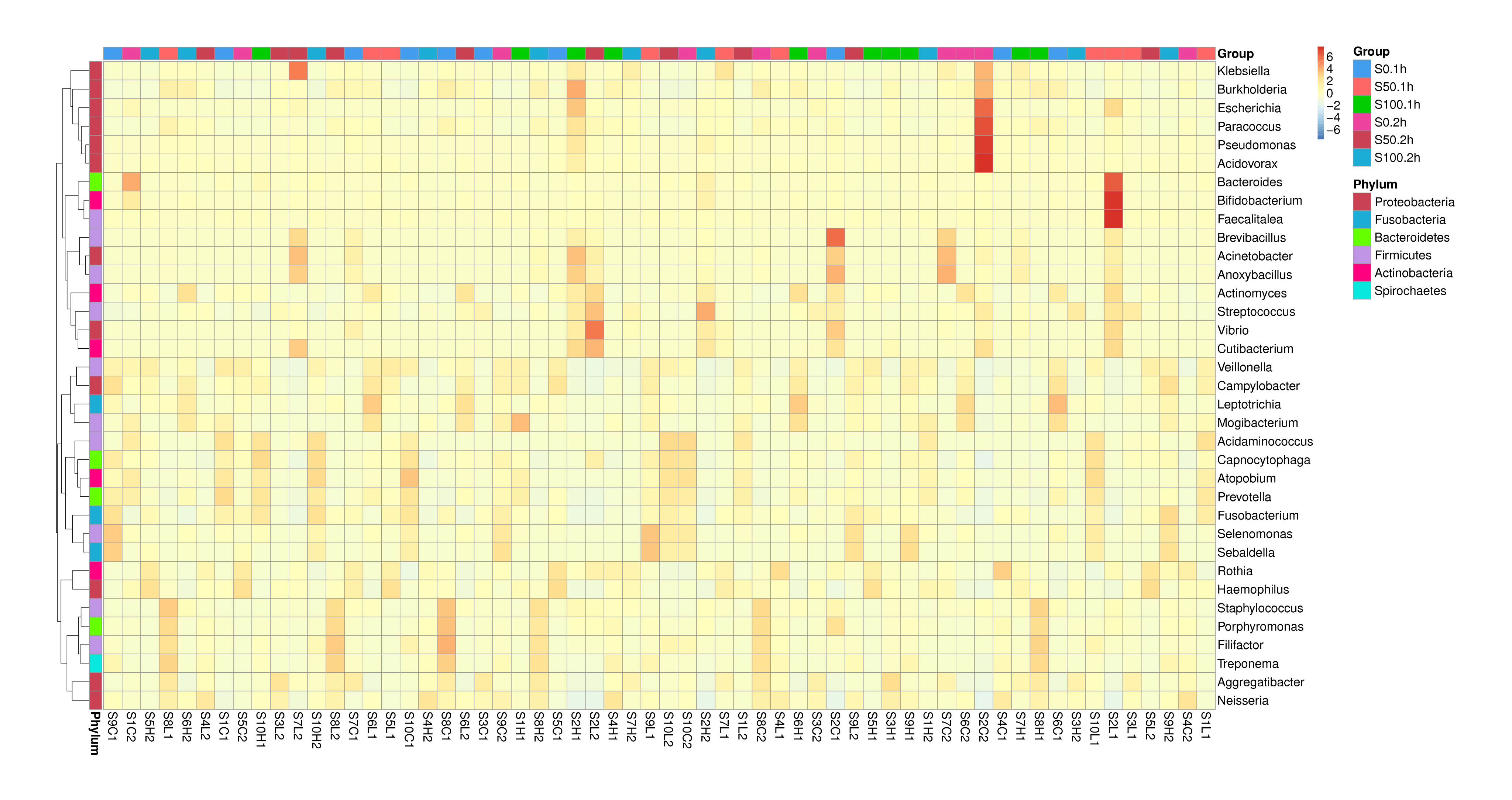
Taxonomic Abundance For Clustering Heatmap
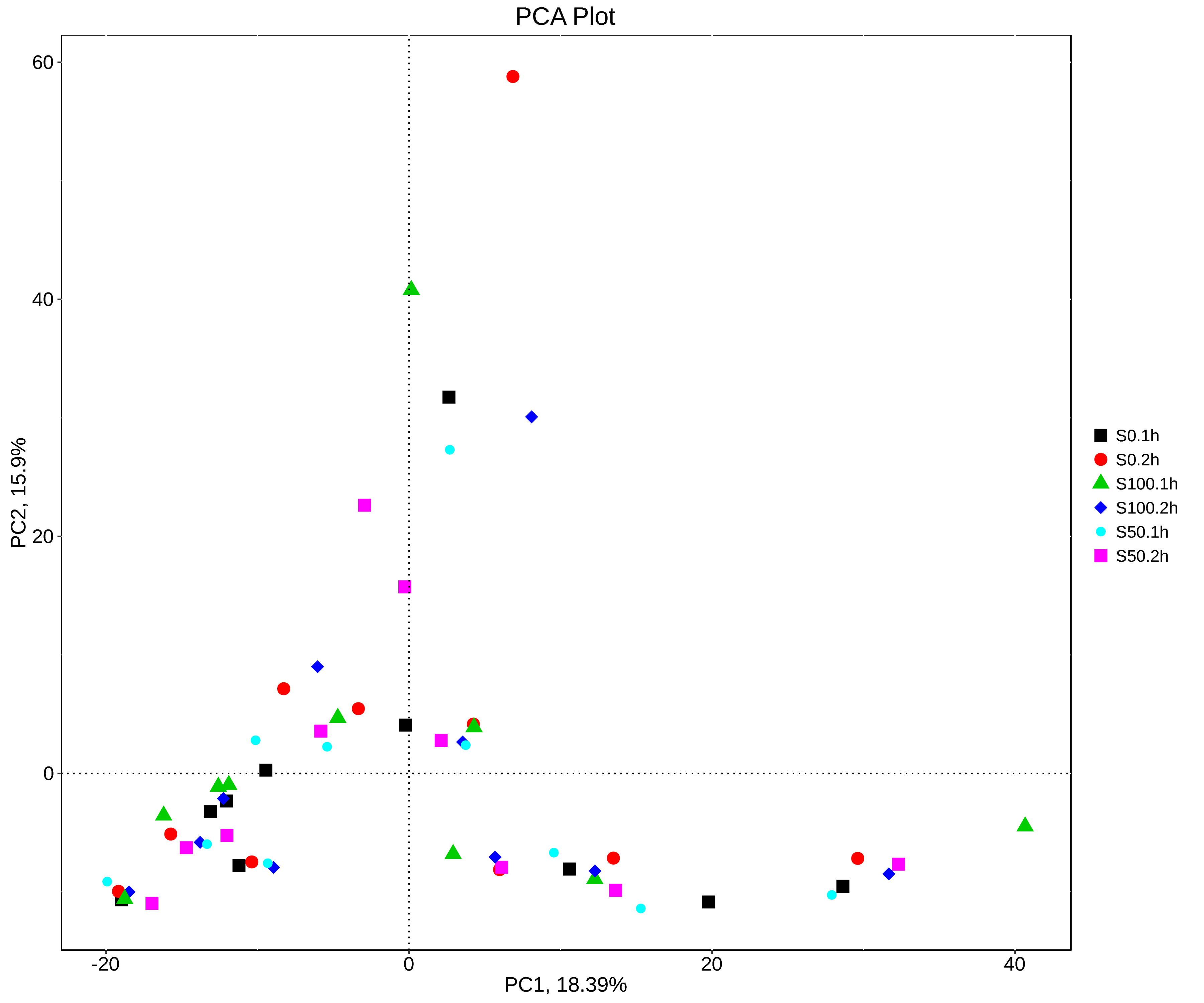
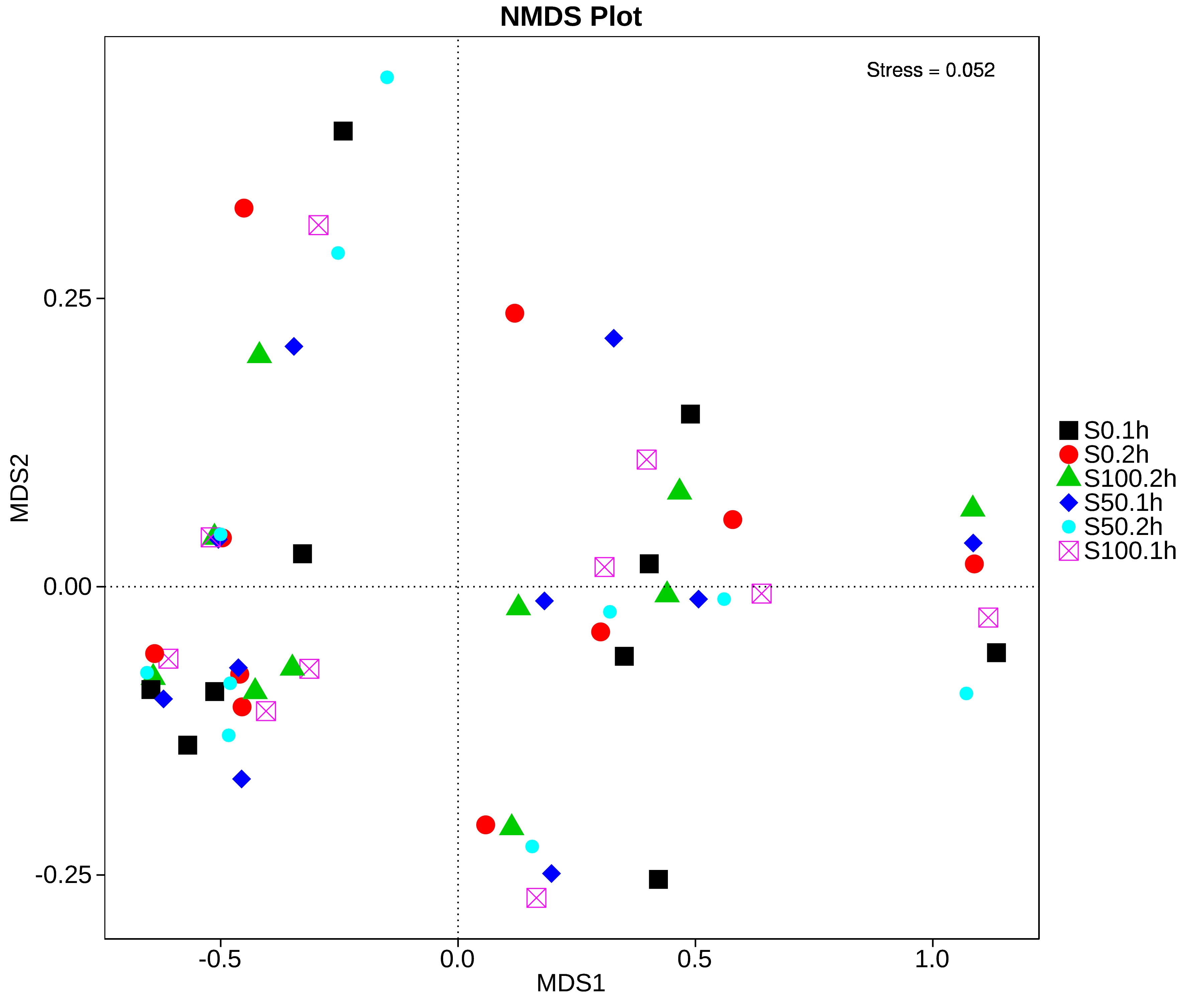
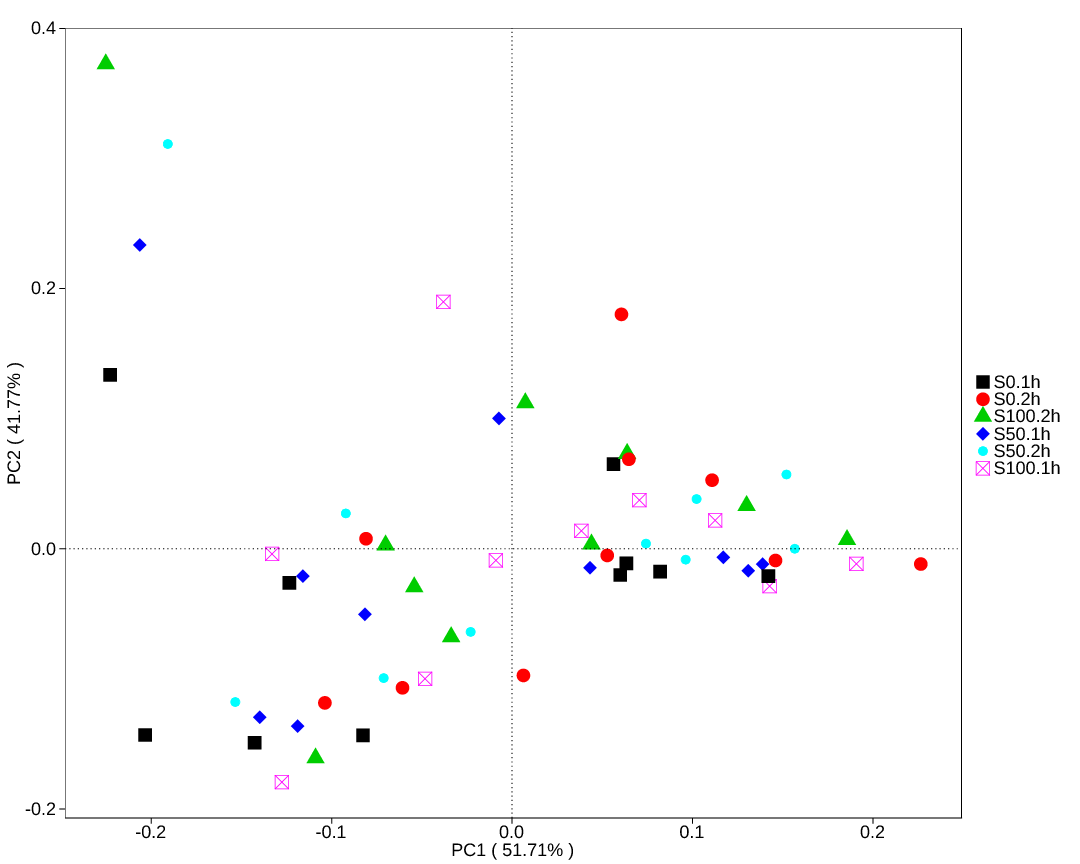
PCA And NMDS Analysis Results Based On Taxonomic Relative Abundance
PCoA Analysis At The Phylum Level Based On Bray-Curtis Dissimilarity
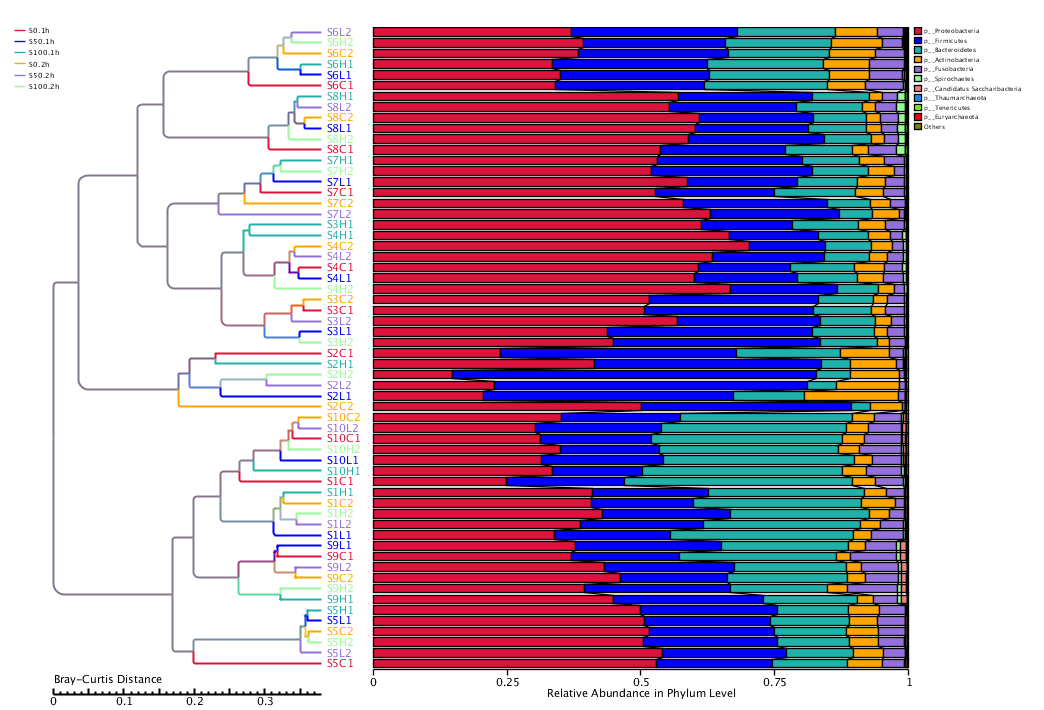
Hierarchical clustering on Taxonomxy Abundance
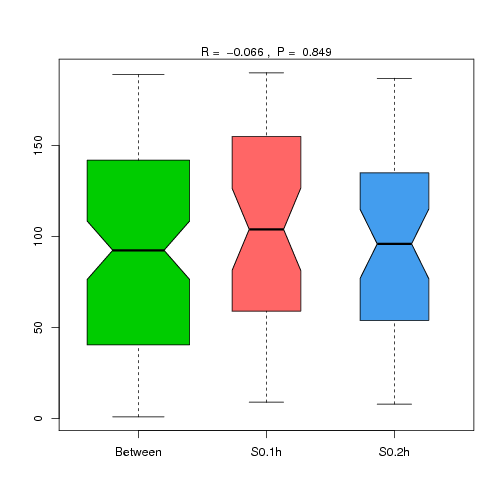
ANOSIM Analysis
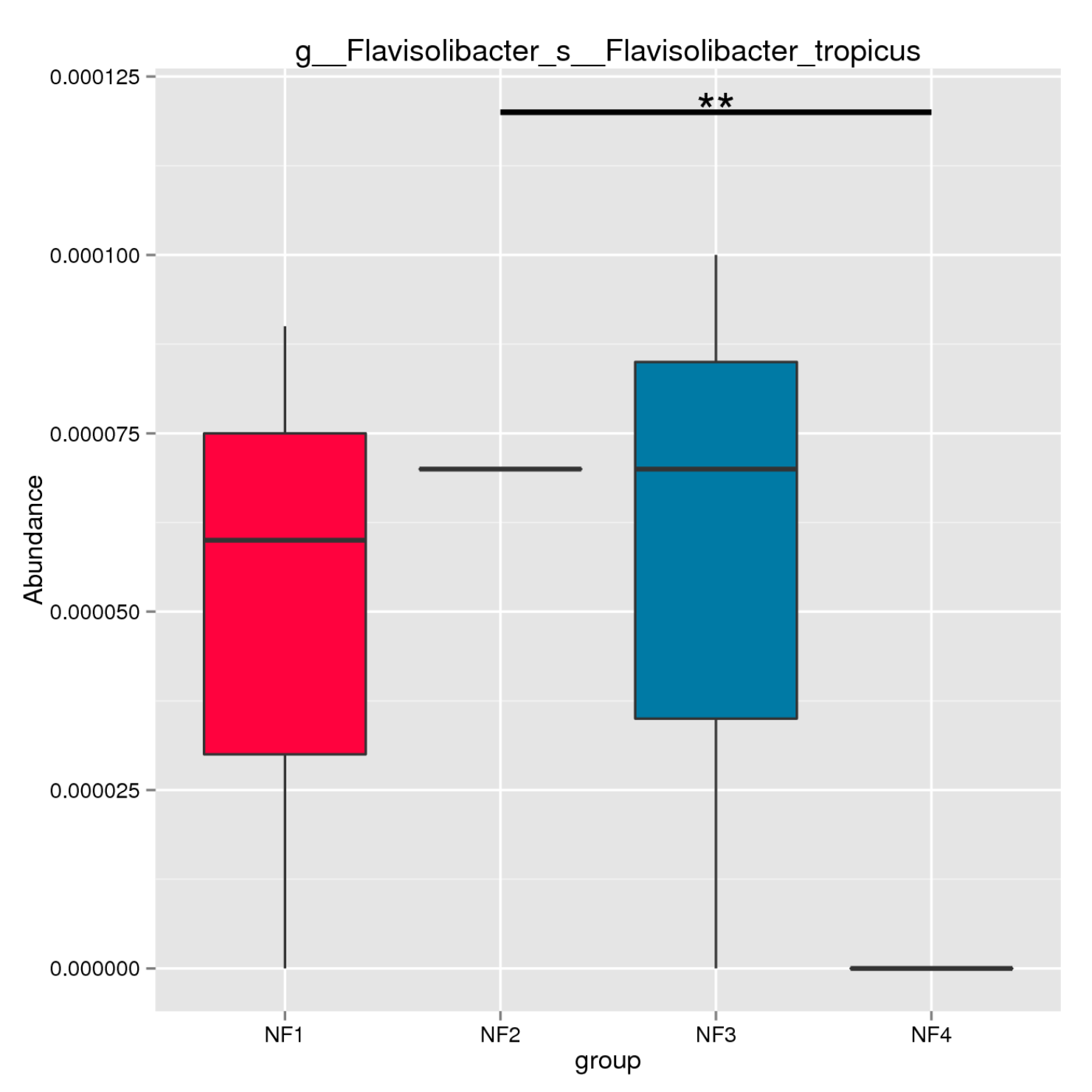
MetaStats Analysis
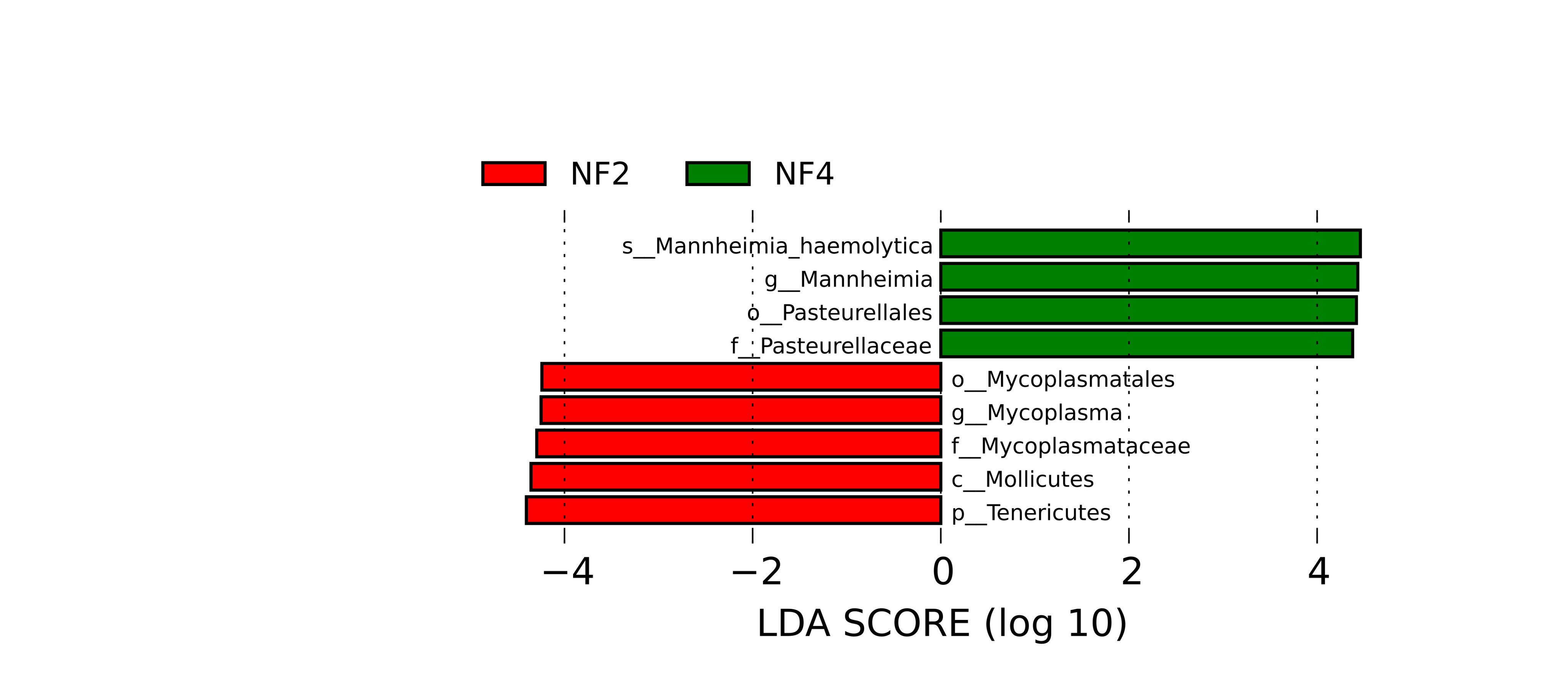
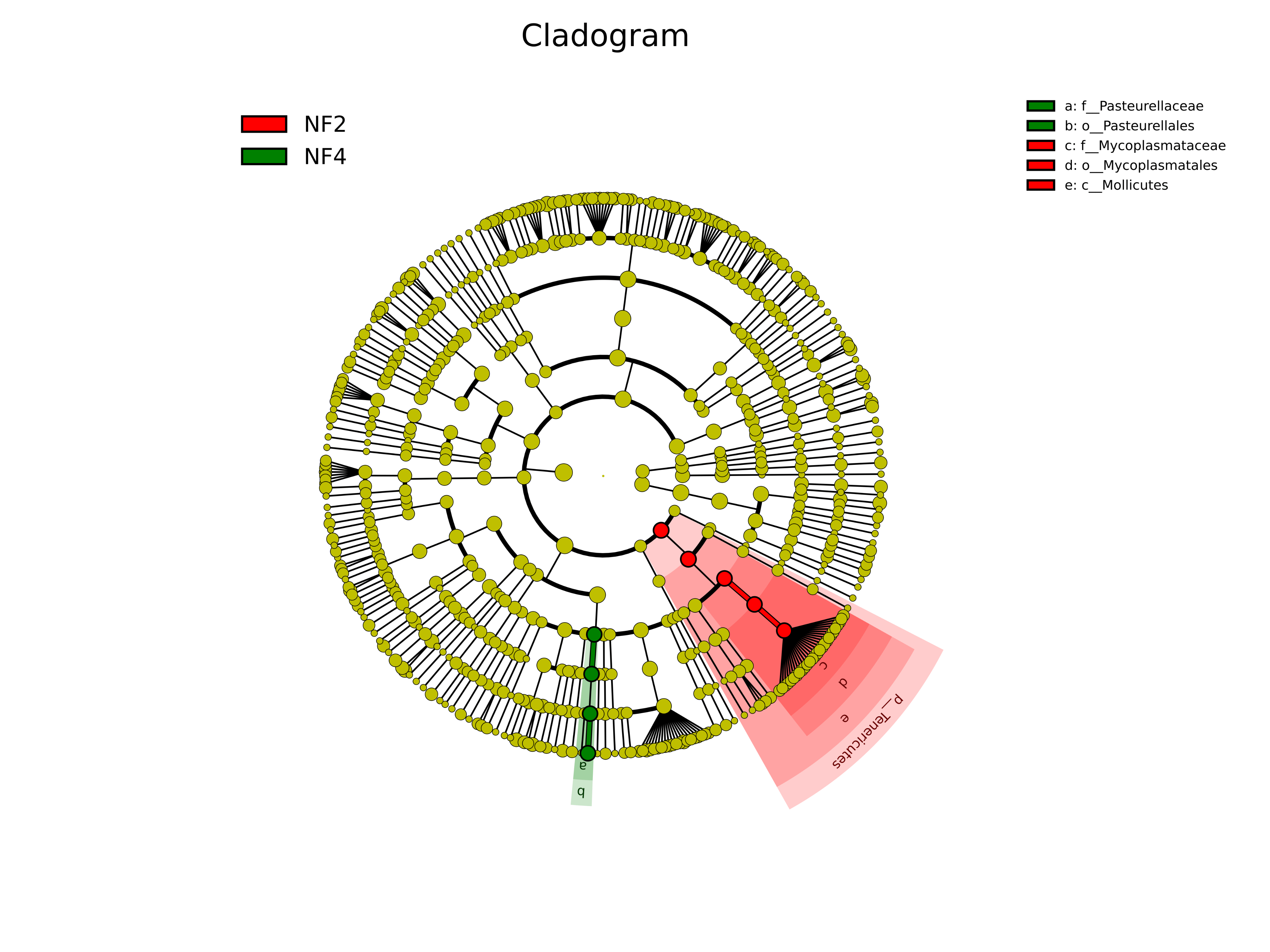
Inter-Group Differences Analysis Using LEfSe Or LDA Effect Size (LEfSe) Analysis