概要

原核生物は主に単細胞生物であり、分類上、膜に結合した核や他のオルガネラを欠いており、転写、翻訳、およびmRNA分解のプロセスがすべて同時に発生する可能性があります。原核生物の転写産物はしばしば複数の遺伝子を含んでおり、複数のタンパク質を指定するポリシストロニックなmRNAを生成する点が、真核生物の転写産物と比較した主な違いです。
原核生物のRNAシーケンスでは、次世代シーケンス(NGS)を使用して、細胞トランスクリプトームの変化を分析することで、特定の瞬間におけるRNAの存在と量を明らかにします。 ストランド特異的RNAライブラリを用いたNovogeneの原核生物RNAシーケンスは、非ストランドRNA-seqと比較して、特にアンチセンスRNAと他の重複遺伝子の両方について、転写産物の発現をより正確に推定することを可能にします。この手法は特に参照ゲノムを持つ原核生物を目的としており、トランスクリプトームプロファイリングや遺伝子構造解析などの費用対効果の高いソリューションをお客様に提供いたします。
リファレンスゲノムのない原核生物で解析する場合、研究目的に合わせてカスタマイズされたサービスも提供できます。詳しくはお問い合わせください。
Service Specificationsアプリケーション
機能ゲノミクス研究
- TSS解析
- プロモーター領域解析
- 5 ‘UTR解析
- オペレーター解析
RNA調節機構の研究
- sRNAの同定
- アンチセンス転写物同定
比較トランスクリプトーム研究
Novogeneの強み
- 数千を超えるプロジェクトを実施した豊富な経験を持ち、いくつもの研究がインパクトファクターの高いジャーナルに掲載されています。
- Illuminaの公式ベンチマークを超えるQ30スコア≥80%が保証する卓越したデータ品質
- 国際的に認められた主流のソフトウェアと成熟した社内パイプラインを使用した包括的なデータ解析を行い、遺伝子発現差を検出し、新規のトランスクリプトを発見し、機能アノテーションを行います。
- Novogeneが提供するソフトウェアはお客様の操作仕様を熟慮したインターフェイスで構成されており、データ解析結果を柔軟に視覚化できます。
サンプル要件
| Library Type | Sample Type | Amount | RNA Integrity Number (Agilent 2100) |
Purity (NanoDrop) |
| Prokaryotic RNA Library | Total RNA | ≥ 3 μg | ≥ 6.0, smooth base line | OD260/280 = 1.8-2.2; OD260/230 ≥ 1.8; |
シーケンスパラメーターと解析内容
| プラットフォームタイプ | Illumin Novaseq 6000 |
| 読み取り長 | ペアエンド150 |
| 推奨されるシーケンス深度 | 参照ゲノムを持つ種について、サンプルあたり2,000万以上のリードペア |
|
標準データ分析
|
データ品質管理 |
| 新規転写産物予測 | |
| 遺伝子発現の定量化と遺伝子発現差プロファイリングと機能解析 | |
| オペロン解析 | |
| SNPおよびInDel | |
| UTR解析 | |
| アンチセンス転写物予測 | |
| sRNA解析 |
注:詳細については、サービス仕様をご参照ください。カスタマイズされたリクエストについては、お問い合わせください。
プロジェクト ワークフロー
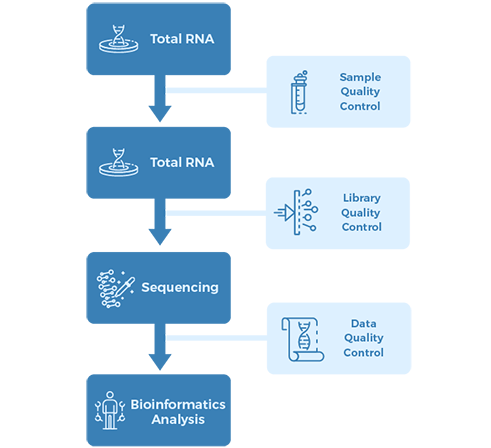
サンプル:
単一細菌コロニーのトータルRNA
シーケンス方法
ライブラリー調製:ストランド特異的RNAライブラリー
シーケンス:HiSeq 2000
結果:
| FunctionI
category |
ORF number in strain 8004(AT33913) | Gene name | Predicted product | Fold change
(hpaR1-/wt) |
Putative HpaR1/Clp co-binding sites |
| Cell envelope and cell structure | XC_1459 (XCC2658) |
phuR | Outer membrane haemin recepton | 2.11 | |
|
Energy and carbon metabolism
|
XC_0279 (XCC0269) |
2,5-Diketo-d-gluconate reductase B | -2.63 | ||
| XC_0281 (XCC0271) |
mocA | Oxidoreductase | -2.80 | GTGTGCGGAACGCTGAATCCACACC | |
| XC_3683 (XCC0549) |
atpE | F0F1 ATP synthase subuni C | -2.56 |
結論:
ここで解析されたデータでは、2つのグローバルな転写レギュレーター、HpaR1とClpがXccの病原性遺伝子のいくつかを共調節する方法を示しています。 RNA-seqは、Xccのトランスクリプトーム全体に対するHpaR1の影響を明らかにしています。
Production of primary metabolites in Microcystis aeruginosa in regulation of nitrogen limitation
緒論:
持続可能なバイオ燃料は多くの注目を集めており、微細藻類は
バイオ燃料生産のための有望な代替原料と考えられています。 多くの研究は異なる微細藻類における炭水化物と脂質の蓄積に焦点を当てていましたが、N欠乏の調節メカニズムを明らかにする報告はそれほど多くありません。 バイオ燃料生産の候補原料である緑膿菌の開発と利用を促進するために、この論文では、さまざまなNレベルでの細胞内の成長、光合成能力、および炭水化物、脂質、タンパク質の含有量を検証し、トランスクリプトームを解析を用いてN欠乏への対応メカニズムを明らかにしました。
サンプル:
Microcystis aeruginosa細胞
シーケンス方法
ライブラリー調製:mRNAライブラリー
シーケンス:Illuminaプラットフォーム
結果:
| Gene ID | Description | Normal-N readcount | Non-N readcount | Fold change Non-N vs. Normal-N | |
|
Photosynthesis
|
MAE_02680 | Precorrin-6y C5, methyltransferase | 121.3±10.7 | 66.3±3.2 | -45.30% |
| MAE_25690 | Precorrin-4 C11, methyltransferase | 34.7±10.3 | 16.7±2.9 | -51.90% | |
| MAE_16230 | Light-independent protochlorophyllide reductase subunit L | 785.3±305.2 | 296.7±85.6 | -62.20% | |
| MAE_61890 | Biliverdin reductase | 114.3±12.3 | 60.3±3.8 | 47.20% |
表2 に N欠乏に応答した炭水化物、Nおよび脂質の光合成および代謝に関連する遺伝子の多様な発現を示しています。
結論:
N欠乏は、Chl合成、アンテナタンパク質、光合成電子伝達系、および細胞の成長に影響を与える炭素固定に関与する遺伝子のダウンレギュレーションを引き起こすことで、緑膿菌の光合成能力を低下させました。 N欠乏状態で蓄積された炭水化物はバイオエタノールの生産に使用できますが、炭水化物抽出後の残りの脂質を抽出することでも、使用可能なバイオディーゼルを生産できます。
エラー率分布
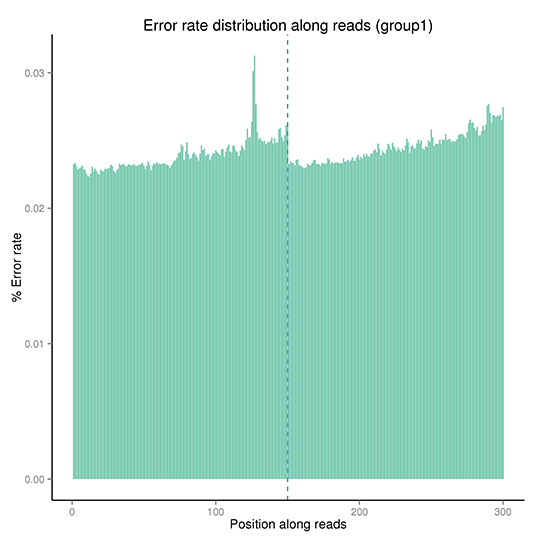
X軸は各シーケンスリードのベース位置を示し、Y軸はベースエラー率を示します。
GC含量の分布
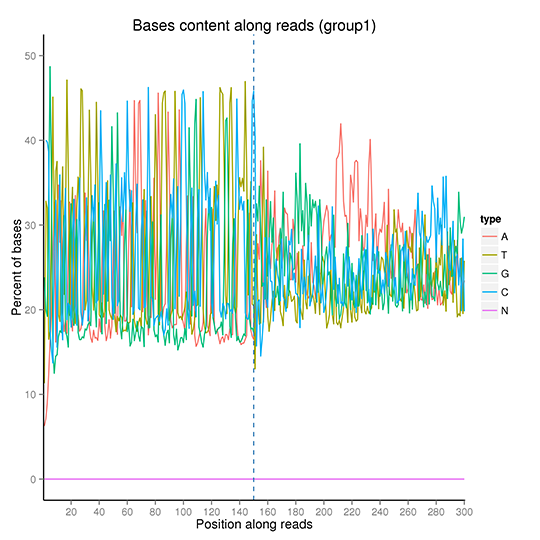
リード位置を横軸に、単一塩基のパーセントを縦軸に示す。 異なる塩基を異なる色で示します。
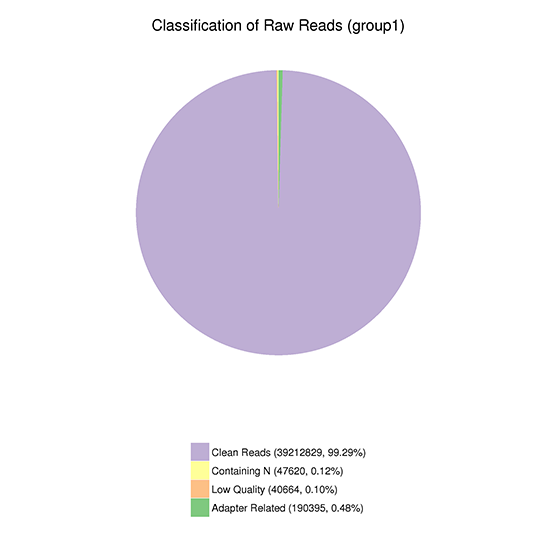
rawデータの構成
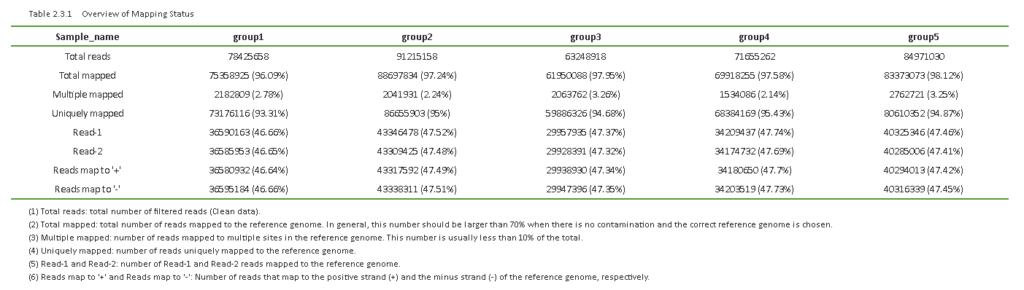
マッピング状況の概要
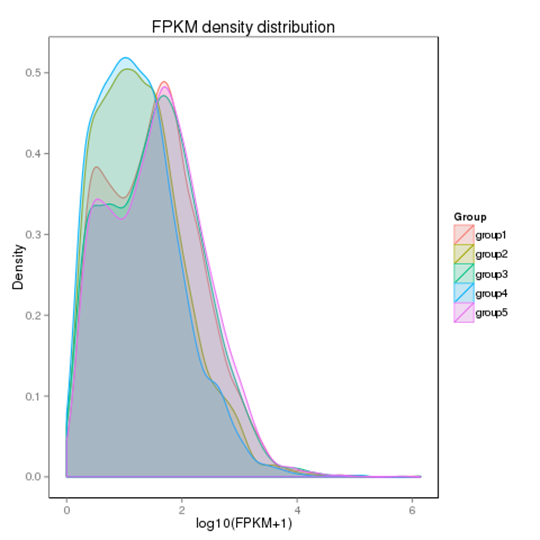
遺伝子発現レベルの分布
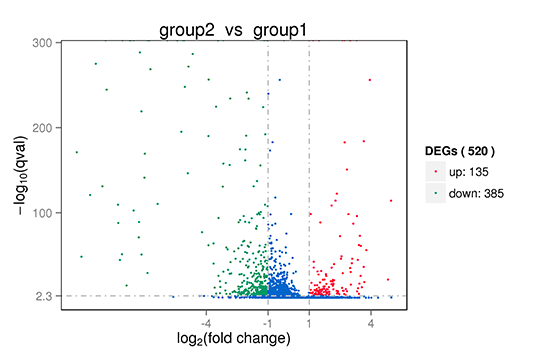
発現差のある遺伝子のVolcano プロット
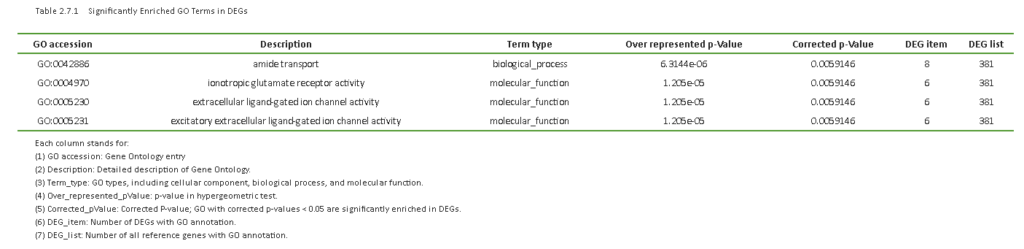
遺伝子発現量の差のうち有意に濃縮したGOパスウェイ
More Research Services