バルクシーケンスと比べてシングルセルシーケンスは、細胞の多様性について完全な複雑性を明らかにします。稀な細胞タイプ、細胞系列の関連性、ヘテロな性質のサンプルの研究に関連する複雑さを解決するために採用されます。詳細なシングルセル解析を通じて、研究者は新しいターゲットやバイオマーカーを効率的に発見し、科学的発見を進展させることができます。
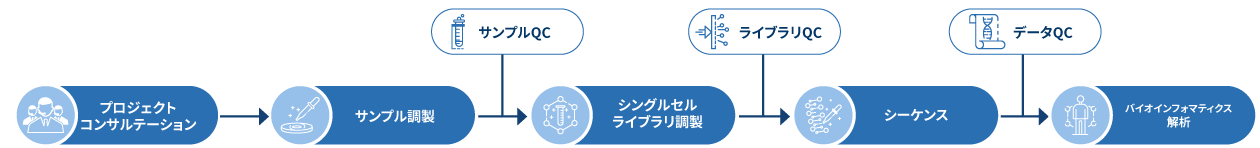
ノボジーンは、包括的な一貫したシングルセルシーケンスサービスを提供しています。ノボジーンのサービスは、実験デザインコンサルティングからオンサイトサービス*、ライブラリ調製、シーケンス、バイオインフォマティクス解析レポートまで、お客様のプロジェクトを包括的にサポートします。
10x GenomicsおよびIllumina NGSプラットフォームの認定サービスプロバイダーとして、当社のシングルセルシーケンシングワークフローにおいて最高品質と信頼性をお約束します。
* オンサイトシングルセル捕捉サービスは、シンガポールと日本でのみご提供しています。
アプリケーション
- シングルセル遺伝子発現: ヘテロな細胞集団から各細胞における細胞の異質性、新規ターゲット、バイオマーカーをプロファイリングします。
- シングルセル免疫プロファイリング: 単一細胞から、全長、ペアB細胞もしくはT細胞レセプター、抗原特異性、および遺伝子発現を解析します。*
- シングルセルATAC: クロマチンアクセシビリティをシングルセルレベルで解析し、細胞の種類や状態、遺伝子制御機構の理解を深めることができます。*
*: AMEA(アジア太平洋、中東、アフリカ)のみで使用可能です。
仕様:サンプル要件
| Sample Type | Service | Sample Amount | Cell Viability |
| Fresh single cell suspensionSingle nuclei suspension* | On-site service* | Single Cells ≥ 1,000,000 | Viability ≥ 80%, Diameter: 5-30 μm |
| Frozen single cell suspension** | Shipping with dry ice | Single Cells ≥ 1,000,000 | Viability ≥ 80%, Diameter: 5-30 μm |
*. オンサイトサービスは、シンガポールと日本でのみご提供しています。
**. 細胞を弊社に送付する場合はフォームをダウンロドし記入したものをご提出ください。
仕様:シーケンスおよび解析
| シーケンスプラットフォーム | イルミナNovaSeq 6000 や NovaSeq X Plus | |
| リード長 | PE 150bp | |
| 推奨データ量 | 20,000 – 50,000 read pairs/cell, around 120Gb/sample | |
| データ解析内容 | Cell Ranger Analysis | Standard Analysis |
|
|
|
仕様:シーケンスおよび解析
お客様が最高品質のデータを得られるよう、一貫したサービススを提供しています。これには、実験デザインに関するアプリケーション専門家とのプロジェクト前のコンサルティング、サンプル準備、ライブラリ構築、シーケンス、データ品質保証、そしてバイオインフォマティクス解析が含まれます。
ノボジーンの標準的な解析パッケージには、異なる細胞集団の特定および遺伝子発現の変動解析が含まれており、研究者に科学的発見を促進するための深い洞察を提供します。Cell Ranger解析パイプラインを活用して、厳密な品質管理、正確なアライメント、および精密な定量化プロセスを通じて、未加工のシーケンスデータを専門的に管理し、複雑なデータを実用的な洞察へと変換します。
ノボジーンの経験は、300を超える種やサンプルタイプにわたり、確立されたモデル生物からシベリアトラ、カメ、ハイギョなどの稀少でユニークな生物システムに至るまで、幅広い生物学的システムを網羅しています。この豊富な経験により、当社は堅牢で信頼性の高い結果を提供することを保証します。
10xシングルセルシーケンスサービスを利用した注目の論文
-
Single-Cell Transcriptome of Wet AMD Patient-Derived Endothelial Cells in Angiogenic Sprouting
Journal: International Journal of Molecular SciencesIssue Date: Oct 2024IF: 6.2DOI: https://doi.org/10.3390/ijms232012549
-
Single-nucleus sequencing deciphers developmental trajectories in rice pistils
Journal: Development CellIssue date: April 06 2023IF: 11.8DOI: https://doi.org/10.1016/j.devcel.2023.03.004
-
Journal: CellIssue Date: Oct 15,2020IF: 31.398DOI: https://doi.org/10.1016/j.cell.2020.08.040
-
Journal: Journal of HepatologyIssue date: Jun 22, 2021IF: 25.083DOI: https://doi.org/10.1016/j.jhep.2021.06.023
-
Journal: Nature CommunicationsIssue date:Jun 23, 2021IF:14.919DOI: https://doi.org/10.1038/s41467-021-24213-6
デモデータ
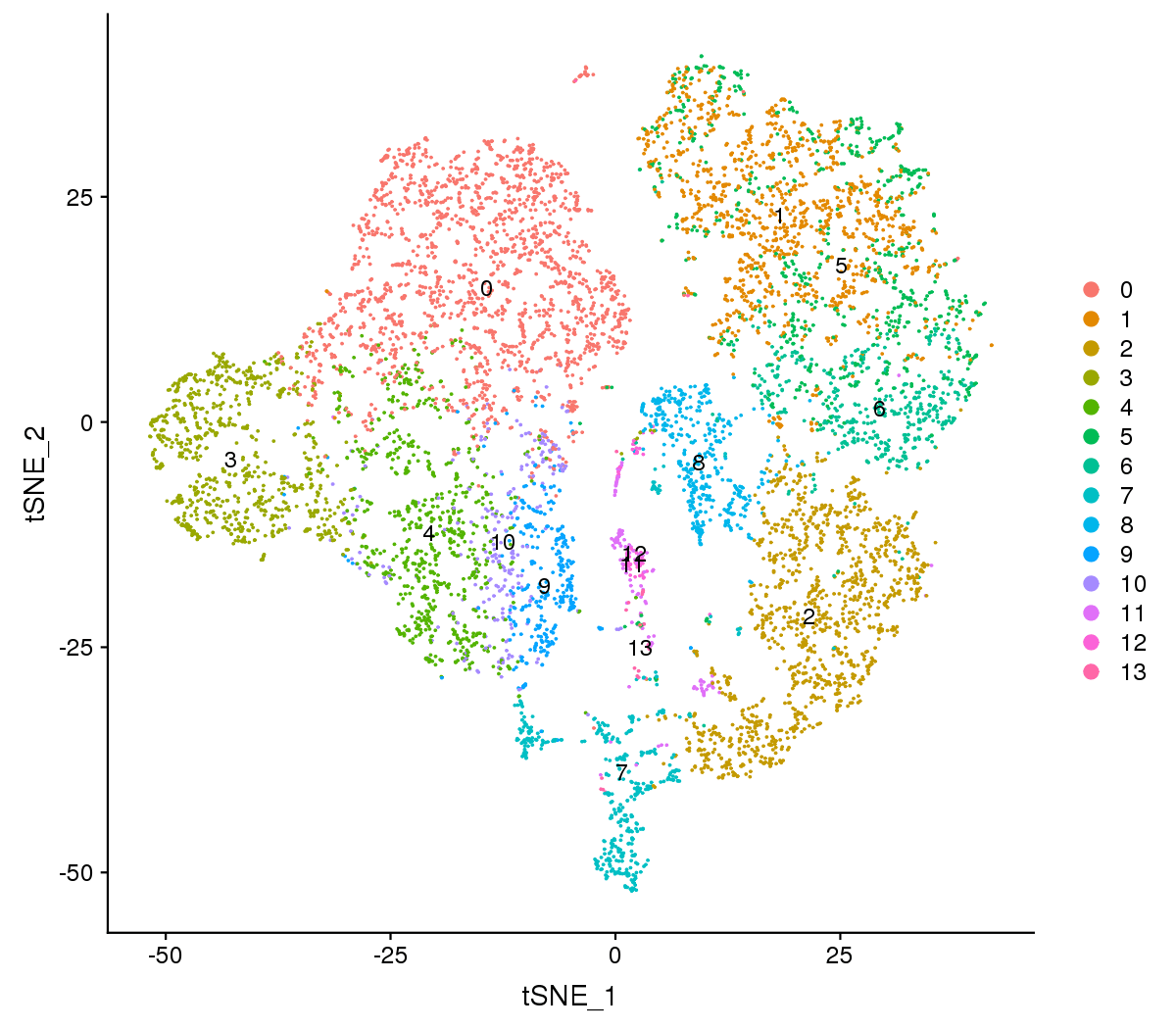
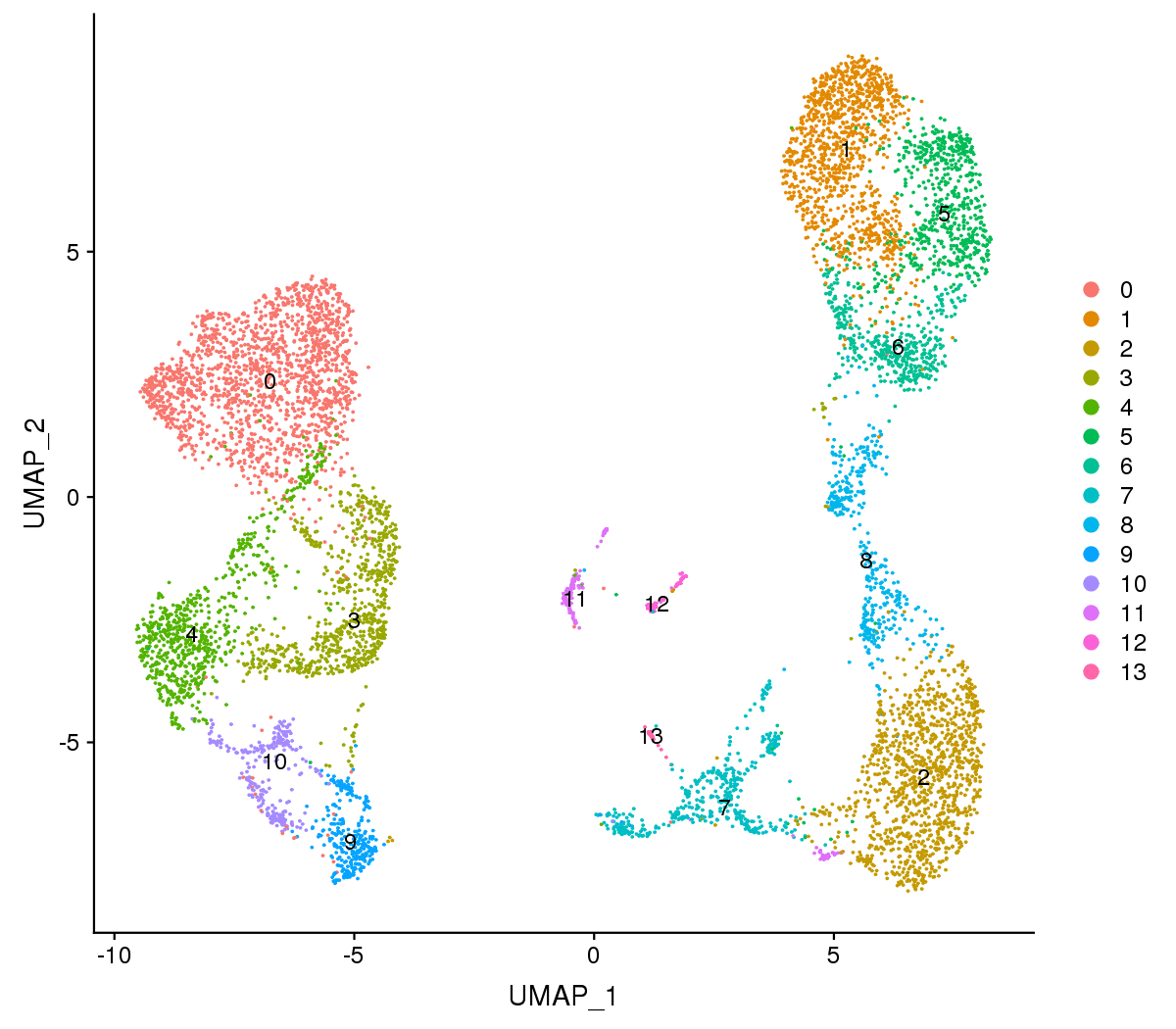
Figure 1 The t-SNE and UMAP plots
Note:Each point represents a cell, and each color designates a cluster.
For visualization purposes, dimensionality was further reduced to 2D using t-distributed stochastic neighbor embedding (t-SNE) and uniform manifold approximation and projection (UMAP). Both of them try to find a low-dimensional representation that preserves relationships between neighbors in high-dimensional space. Compared to t-SNE, the UMAP visualization tends to have more compact visual clusters with more empty spaces between them.
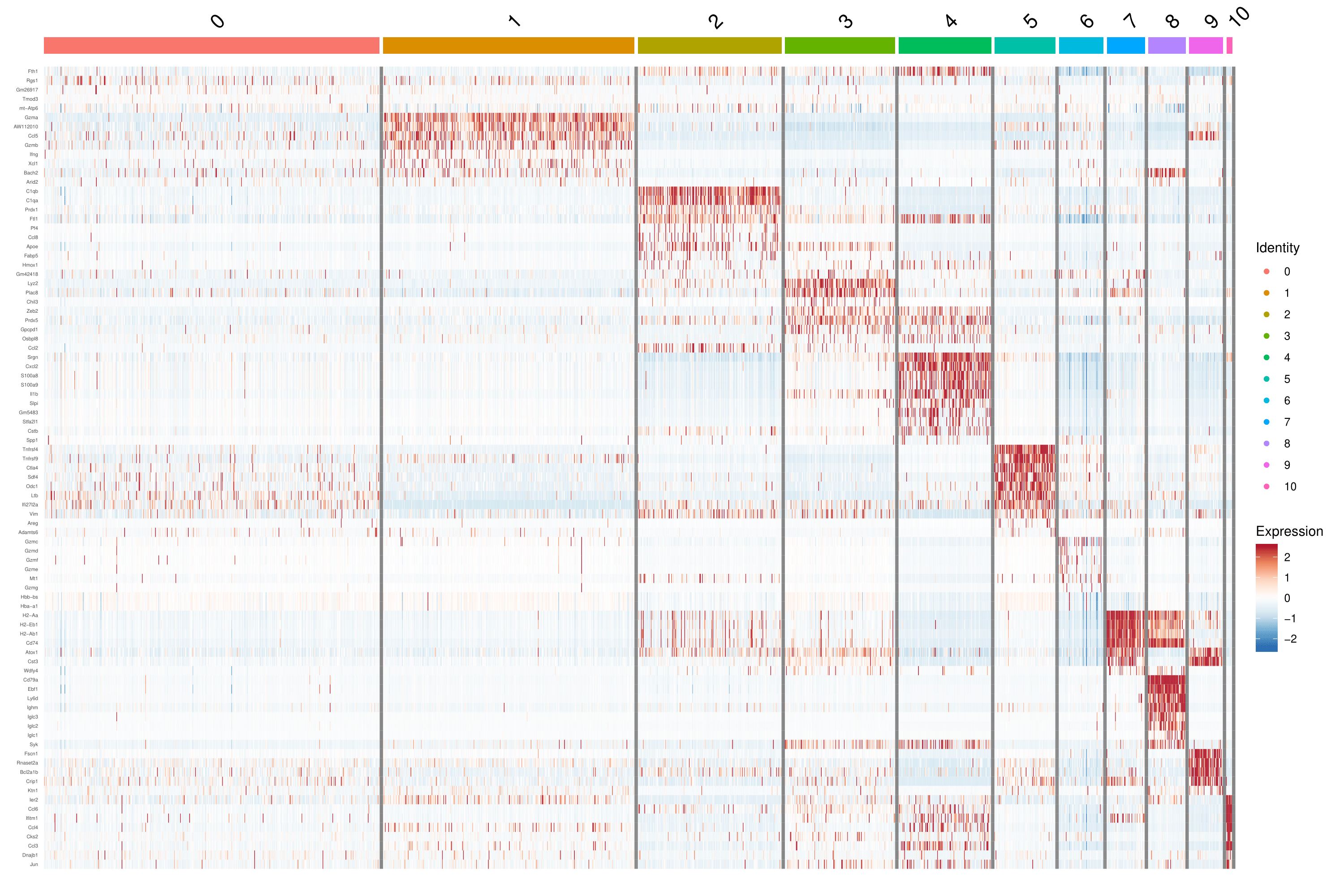
Figure 2 Expression Pattern of Marker Genes
Note: Darker red represents a higher expression level, and darker blue dictates a lower expression level.
Heatmaps show the expression of indicated marker genes for given cells and features. In this case, we plotted the top 10 markers (or all markers if less than 10) for each cluster.
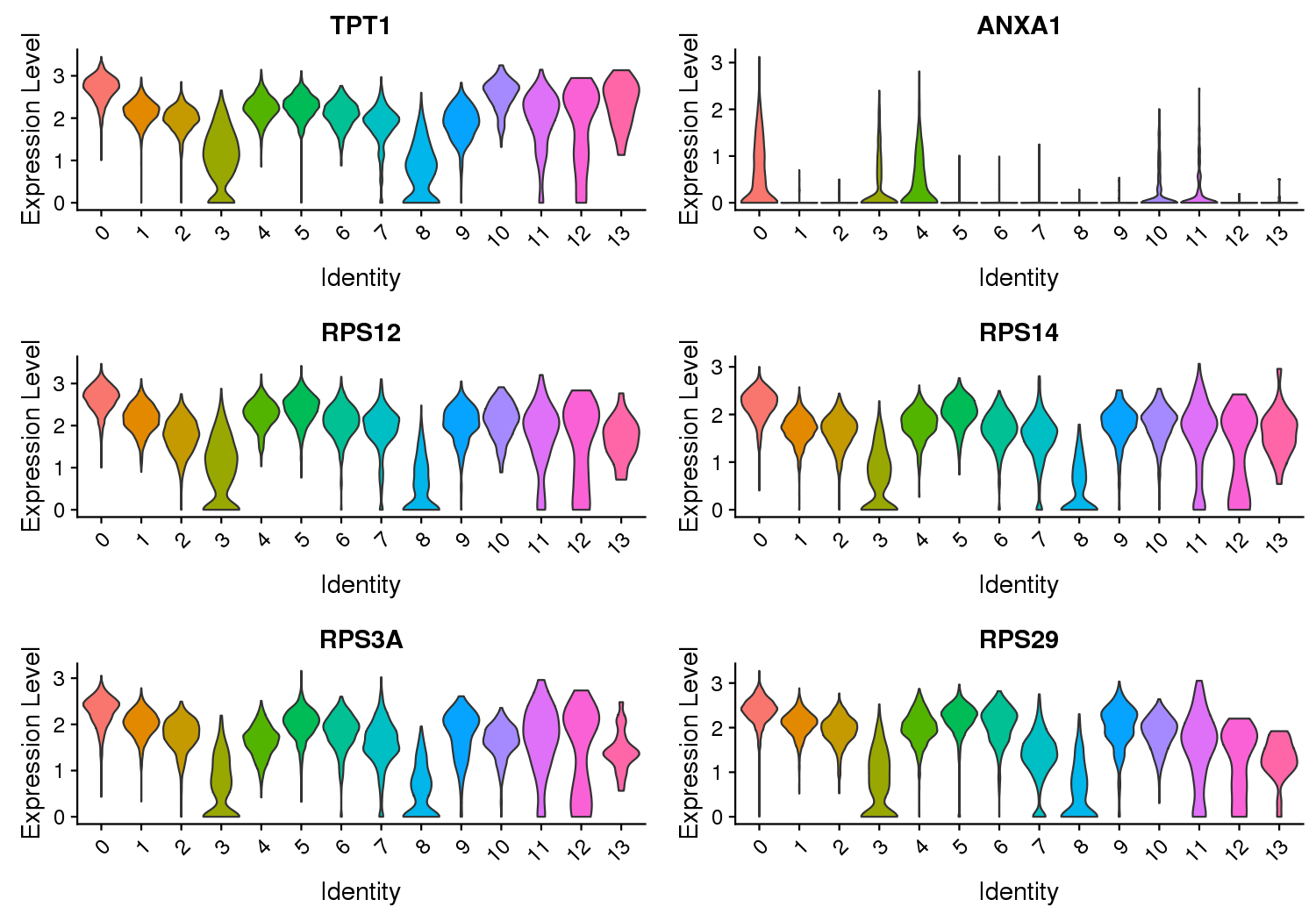
Figure 3 Expression Level of Marker Genes
Note: The X-axis represents the cluster, and the Y-axis represents the expression level.
Violin plots show the relative expression levels of marker genes among all clusters. (Only six marker genes are shown here)
その他のサービス