概要

メタトランスクリプトームとは、自然界の微生物群集(すなわち、土壌、水、海、糞、腸)における遺伝子転写産物(遺伝子のRNAコピー)の総量を指し、それらはサンプリング時固有の環境状況の影響を受けます。メタトランスクリプトームは時間的および環境的変動によって変化します。次世代シーケンス(NGS)を用いてメタトランスクリプトームシーケンス解析を行うことで、群集内の発現プロファイルの全体像を取得し、経時的または環境パラメータによる遺伝子発現パターンのダイナミクスを追跡することで、複雑な微生物叢における、構造、機能、および複雑適応メカニズムの理解を深めることができます。
メタトランスクリプトームシーケンスは、真核生物と原核生物の両方の微生物の自然環境内での遺伝子発現を同定します。具体的には、このサービスを使用すると、複雑な微生物群集の遺伝子発現プロファイリング全体、種の分類学的分析、さまざまに発現した遺伝子の機能パスウェイ解析などを取得できます。 Novogeneでは、慎重な解析前検証を提供することで、お客様のプロジェクトに合わせて調整を行い、研究目標をより容易に実現するようサポートいたします。
Service Specificationsアプリケーション
微生物生態学研究:
- 機能性活性細菌の特性評価
- コミュニティの代謝相互作用
臨床研究:
- Immune recognition
- Immune education
- 自己免疫疾患
バイオテクノロジー研究
Novogeneの強み
- 数千のサンプルのシーケンスを実施した豊富な経験。
- Illuminaの公式ベンチマークを超えるQ30スコア≥80%が保証する卓越したデータ品質
- 国際的に認められた主流ソフトウェアと成熟した社内パイプラインを使用した包括的な解析を実施し、広範なバイオインフォマティクスの需要に対応します。
サンプル要件
| Library Type | Sample Type | Amount | RNA Integrity Number (Agilent 2100) |
Purity« (NanoDrop) |
|
Meta-transcriptome Library
|
Total RNA
|
≥ 2.5 μg
|
≥ 6.5, smooth base line
|
OD260/280 = 1.8-2.2;
OD260/230 ≥ 1.8; |
シーケンスパラメーターと解析内容
| プラットフォームタイプ | Illumin Novaseq 6000 |
| 読み取り長 | ペアエンド150 |
| 推奨されるシーケンス深度 | 参照ゲノムを持つ種について、サンプルあたり4,000万以上のリードペア |
|
標準データ分析
|
|
注:詳細については、サービス仕様をご参照ください。カスタマイズされたリクエストについては、お問い合わせください。
プロジェクト ワークフロー
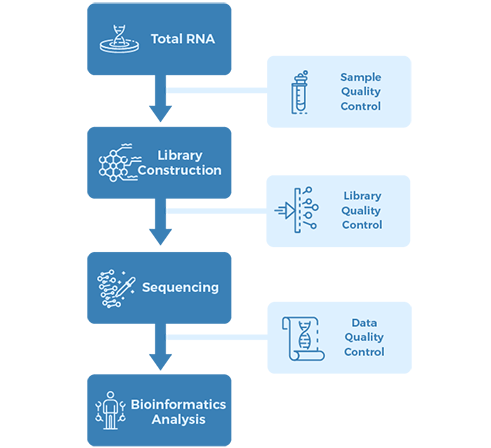
サンプル:
ロングアイランド島の水
シーケンス方法
ライブラリー調製:ストランド特異的RNAライブラリー
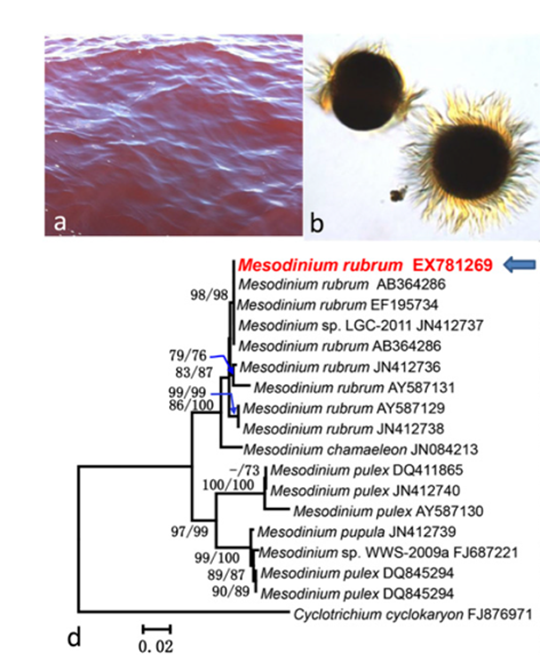
(A)赤潮の写真。
(B)原因となる繊毛虫の光学顕微鏡像。
(C)原因となる繊毛虫がM. rubrumであることを証明する18S rDNA系統樹。

結論:
Mesodinium-farming-Teleaulaxモデルは、現在のMesodiniumに隷属化された葉緑体またはオルガネラ複合体の現在のモデルとは区別されるだけでなく、共生細胞の増殖を促進することを示し、ホストの分裂と同期することが考えられている、分裂のみが同期する従来の細胞内共生体モデルとは異なります。それぞれの場合の生態学的重要性は、今後さらに研究されるべきです。
図1 エラー率分布
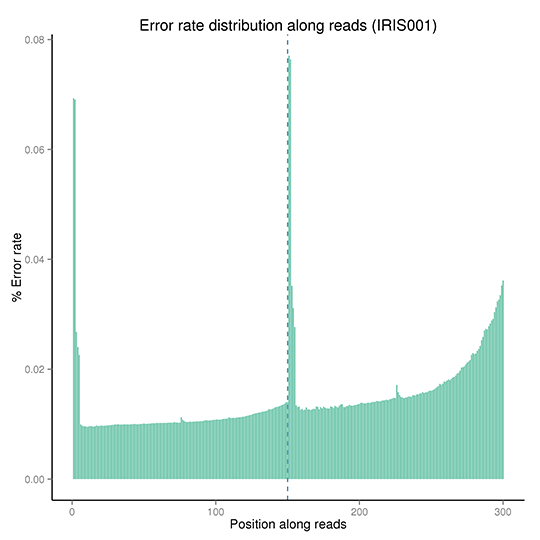
X軸は各シーケンスリードのベース位置を示し、Y軸はベースエラー率を示します。
図2 GC含量の分布
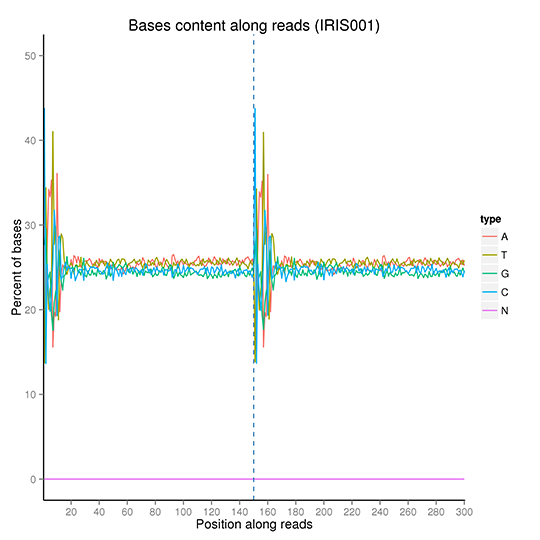
リード位置を横軸に、単一塩基のパーセントを縦軸に示す。 異なる塩基を異なる色で示す。
図3 rawデータの構成
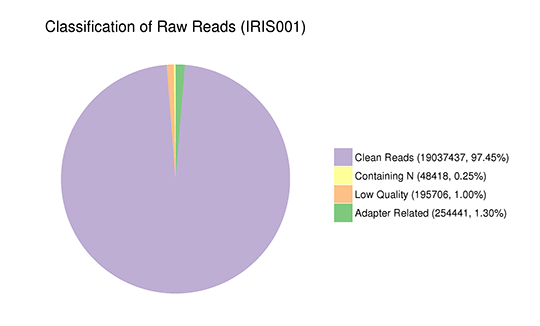
図4 転写産物と単一遺伝子の長さの分布
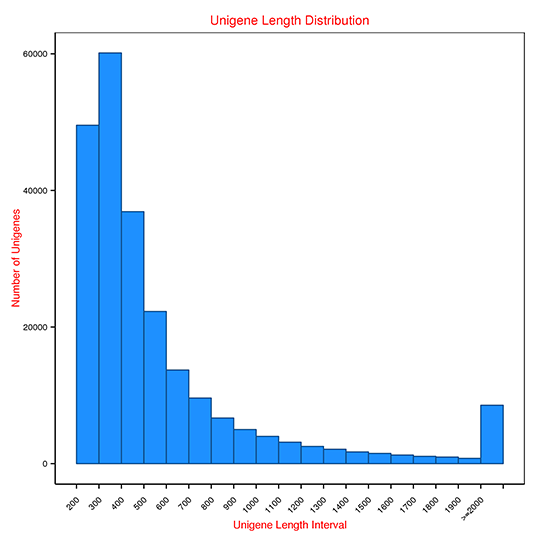
図5 種の多様性のクラスタリング。
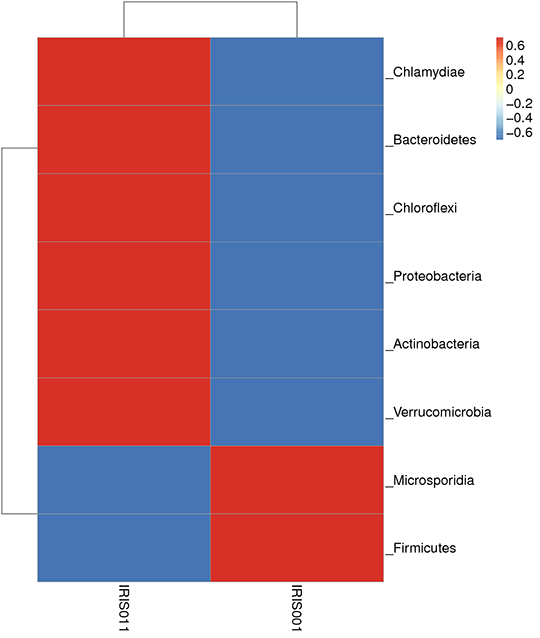
図6 転写物の定量。
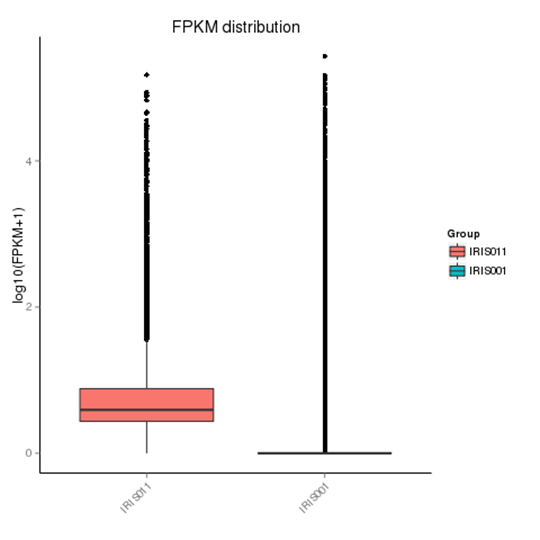
図7 発現差のある遺伝子のVolcano プロット
More Research Services