マウス全エクソームシーケンスの紹介
マウス全ゲノムシーケンス(mWES)は、全ゲノムシーケンス(WGS)よりも比較的費用対効果の高い手法であり、変異の同定のためにマウスゲノムのエクソン領域をターゲットとすることを目的としており、膨大な変異体グループのハイスループット遺伝子解析に特に有益です。エクソームは全ゲノムのわずか1%に過ぎないため[1]、マウス全ゲノムシーケンスでは、WGSと比較して非常にコンパクトで少ないデータで、より詳細なシーケンスに関する知見を得ることができます。
ノボジーンでは、マウス全エクソームシーケンスのDNA抽出、ライブラリー調製、シーケンス、解析を含むエンドツーエンドのサービスプロセスを提供しています。これらのサービスにより、研究者は特徴的な表現型を持つマウスの原因変異を同定し、ヒト疾患に関連する遺伝子を発見し、マウスモデルにおける薬剤耐性進化のメカニズムを明らかにすることができます。
マウス全エクソームシーケンスの応用例
マウス全エクソームシークエンシングは、研究者が以下のメカニズムを解明する上で役立っています:
- がん治療における薬剤開発
- ヒトの健康と疾患に関するモデル研究
ノボジーンのマウス全エクソームシーケンスの利点
- mWES サービスでは、Agilent SureSelectXT Mouse Exon Kit および NovaSeq 6000 プラットフォームをご利用頂けます。
- Q30≥85%を保証するデータ品質。
- mWESにおける標的領域の平均カバレッジは99.6+/-。
- ノボジーンの最先端のバイオインフォマティクス・パイプラインとデータベースに基づき、結果概要のための洞察に導く論文に用いられうるデータと図を提供します。
mWES仕様:サンプル要件
| サンプルタイプ | サンプル量 (Qubit®) | 純度 |
| Genomic DNA | ≥ 300 ng | A260/280=1.8-2.0; no degradation, no contamination |
| Genomic DNA from FFPE tissue | ≥ 500 ng | Fragments should be longer than 1000 bp |
mWES仕様:シーケンスと解析
| シーケンスプラットフォーム | Illumina NovaSeq 6000 |
| リード長 | Paired-end 150 bp |
| 推奨シーケンス深度 | Effective sequencing depth above 50× (6G) |
| 標準データ解析内容 |
|
注:詳細情報については、サービス仕様をご参照ください。カスタマイズされたご要望についてはお問合せください。
ノボジーン mWESサービスワークフロー
データQCシーケンスエラー率分布
シーケンスエラー率は、ディープシーケンスによる低頻度変異の正確な検出の主要な交絡因子です。これはシーケンスデータの質を決定します。シーケンスエラー率は、シーケンスサイクルと大きく関連しており、イルミナのハイスループットシーケンスプラットフォームの一般的な特徴である試薬の消費により、各リードの終わりに向かって上昇します。
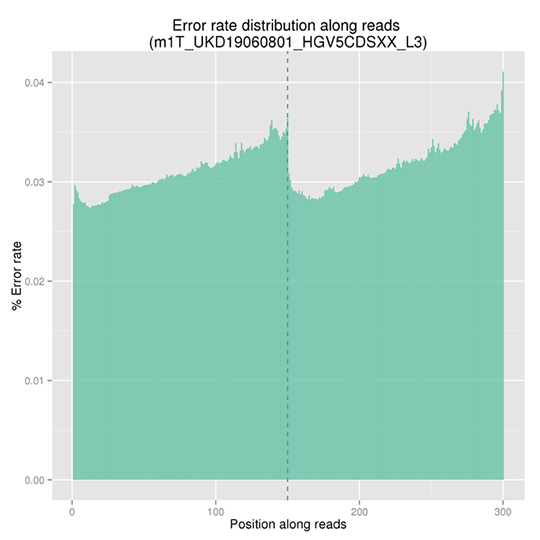
注:X軸はリードの位置を表し、Y軸はその位置における全リードの平均塩基エラー率を示します。
GC含量分布
GC含量分布は、AT/GC分離の可能性をチェックすることを目的としています。サンプルのコンタミネーション、シーケンスバイアス、ライブラリー調製時のエラーは、シーケンス結果に影響を与える可能性があります。
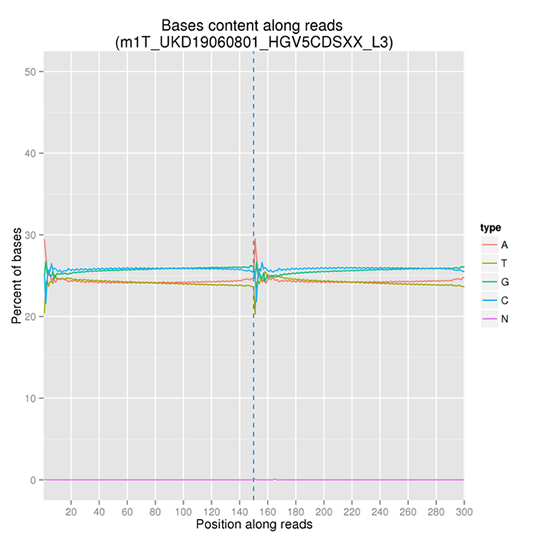
注:X軸はリードの位置を表し、Y軸は各塩基の種類(A、T、G、C)の割合を示します。異なる塩基は異なる色で区別できます。
参照ゲノムとのアラインメントシーケンスデプスとカバレッジの分布
シーケンスデプスとカバレッジは、既知の参照ヌクレオチドにアライメントされたペアエンドクリーンリードの平均数を示しています。シーケンスカバレッジ分布は、特定の塩基位置で変異の同定がある程度の信頼性をもって行えるかどうかを決定します。
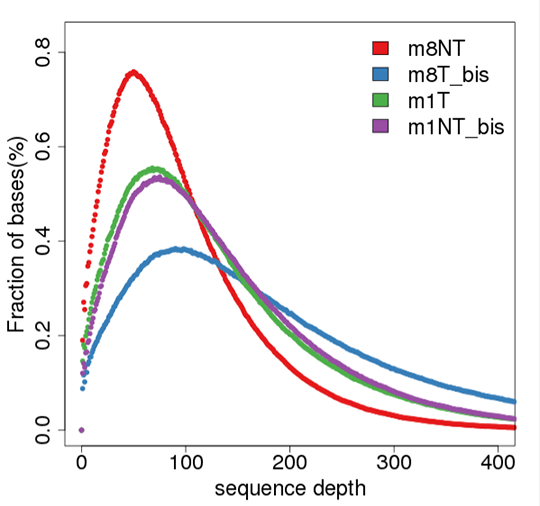
注:図は各サンプル中の全塩基のシーケンスデプス分布を示しています。X軸はシーケンスデプス、Y軸は指定されたシーケンスデプスの塩基の割合です。
SNPおよびInDelコール, アノテーションおよび統計
一塩基多型(SNPs)は、一塩基変異体(SNVs)としても知られ、ゲノム中の遺伝子バリアントの最大のクラスを構成します。遺伝的変異のもう一つのクラスは、長さが50bp未満の小さな挿入と欠失(InDels)です。
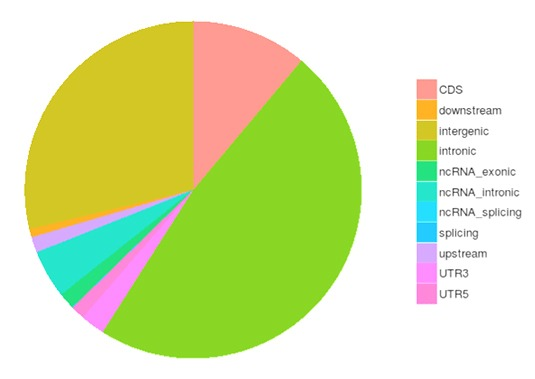
様々なゲノム領域におけるSNPs/lnDelsの数
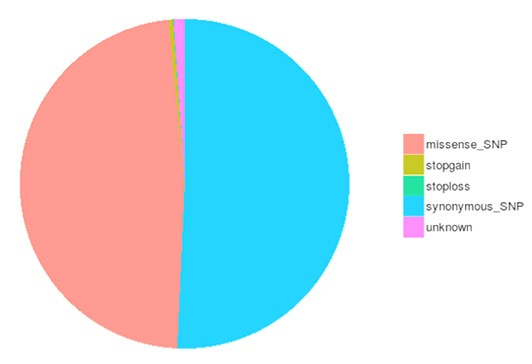
コーディング領域のSNPs/InDelsの異なるタイプ数
*フルバージョンのデモレポートをご希望の方はお問い合わせください。
その他の解析サービス