概要
16S/18S/ITS アンプリコンメタゲノムシーケンスは、特定の領域(アンプリコン)をターゲットとして詳細な解析を行うDNAシーケンス手法です。このアプローチでは、PCRを用いて保存遺伝子または遺伝子間領域の短い(<500 bp)超可変領域を増幅し、次世代シーケンス(NGS)を行います。この手法により、複雑なサンプル内の複数の微生物種の同定と区別が可能になります。アンプリコンメタゲノムシーケンスは、細菌および古細菌の16SリボソームRNA(rRNA)遺伝子、真核生物の18S rRNA遺伝子、および真菌(酵母、カビ、およびその他の関連種を含む)の内部転写スペーサー(ITS)領域を特異的にターゲットとしています。これにより、包括的な系統学的および分類学的分析が可能になります。
ノボジーンのアンプリコンメタゲノムシーケンスサービスは、ショットガンメタゲノムシーケンスと比較してコスト効率の高いアプローチで標的生物をスクリーニングし、複雑な微生物群集の多様性に関する知見を提供します。このアプローチは、微生物生態学研究、特定の微生物群の系統解析、純粋培養における種の同定、病原性か有益性かにかかわらず関心のある生物の検出などに広く利用されています。
16S/18S/ITS アンプリコンメタゲノムシーケンスはどのように機能するのか?
アンプリコンメタゲノムシーケンスは、多様なサンプル中の微生物の全スペクトルを探索するための強力な手法です。この手法は特に次のような場合に有効です:
- 複雑な微生物集団の中から目的の特定の生物をターゲットとし、同定する。
- 微生物群集の包括的な側面を提供するために、多種多様な微生物に注釈を付け、分類する。
- 正確な分類学的解析のために、細菌や古細菌(16S)と、真菌や原生生物(ITS/18S)のような他の真核生物を区別する。
- 病原体、微生物汚染、有益な土壌細菌、その他の関連微生物の検出。
- 生理的流体や組織サンプルを分析し、疾患や癌の診断に役立てる。
このアプローチは、微生物の多様性に関する詳細な洞察を提供し、様々な研究や診断アプリケーションにおける貴重なツールとなります。
利点
ノボジーンは、アンプリコンシーケンスプロジェクトにおける豊富な経験を活かし、近年、アンプリコンメタゲノムシーケンス用のショートリードおよびロングリード(16S Full-Length Amplicon Sequencing)技術を含む最先端のパイプラインを開発しました。
また、Qiime1パイプラインによるOTU(Operational Taxonomic Units)へのクラスタリングや、Qiime2パイプラインのDADA2によるASV(Amplicon Sequence Variants)へのノイズ除去など、ノボジーンのサービスは、実験デザインにおいて比類のない柔軟性を提供します。さらに、Qiime 1パイプラインとQiime 2パイプラインの両方を通じて、カスタマイズされた解析とデータベースを備えたテーラーメイドのソリューションを提供し、特定の研究要件との正確な適合を保証します。
アンプリコンメタゲノムシーケンスに用いられるプライマー
| タイプ | 増幅領域 | フラグメント長 | プライマー | 配列(5’-3’) |
| 細菌16S | V4 | 300 bp | 515F | GTGCCAGCMGCCGCGGTAA |
| 806R | GGACTACHVGGGTWTCTAAT | |||
| V3-V4 | 470 bp | 341F | CCTAYGGGRBGCASCAG | |
| 806R | GGACTACNNGGGTATCTAAT | |||
| V4-V5 | 450 bp | 515F | GTGCCAGCMGCCGCGGTAA | |
| 907R | CCGTCAATTCCTTTGAGTTT | |||
| V5-V7 (for endophytic) | 435 bp | 799F | AACMGGATTAGATACCCKG | |
| 1193R | ACGTCATCCCCACCTTCC | |||
| 古細菌 V4 (AKA 新規古細菌 V4) |
V4-V5 | 415 bp | Arch519F | GTGCCAGCMGCCGCGGTAA |
| Arch915R | GTGCTCCCCCGCCAATTCCT |
| タイプ | 増幅領域 | フラグメント長 | プライマー | 配列(5’-3’) |
| 糸状菌 18S | V4 | 350 bp | 528F | GCGGTAATTCCAGCTCCAA |
| 706R | AATCCRAGAATTTCACCTCT | |||
| 糸状菌 ITS | ITS1-5F | 200-400 bp | 1737F | GGAAGTAAAAGTCGTAACAAGG |
| 2043R | GCTGCGTTCTTCATCGATGC | |||
| ITS2 | 320-380 bp | ITS3 | GCATCGATGAAGAACGCAGC | |
| ITS4 | TCCTCCGCTTATTGATATGC | |||
| ITS1-1F (for endophytic) |
200-400 bp | ITS1F | CTTGGTCATTTAGAGGAAGTAA | |
| ITS2 | GCTGCGTTCTTCATCGATGC |
仕様:DNAサンプル要件
| サンプルタイプ | サンプル量 | 液量 | 濃度 | 純度 |
| total DNA | ≥ 200 ng | ≥ 20 μL | ≥ 10 ng/μL |
OD260/280 = 1.8-2.0
分解なし, 色なし, コンタミなし |
仕様:シーケンスと解析
| シーケンス プラットフォーム |
イルミナNovaSeq 6000 | |
| シーケンス法 | NovaSeq 試薬 (500サイクル) | |
| Data Output | 50K/100K/500K raw tags | |
| Qiime1/Qiime2 | 標準解析 |
|
| 高次解析 |
|
|
アンプリコンメタゲノムシーケンスを用いた論文
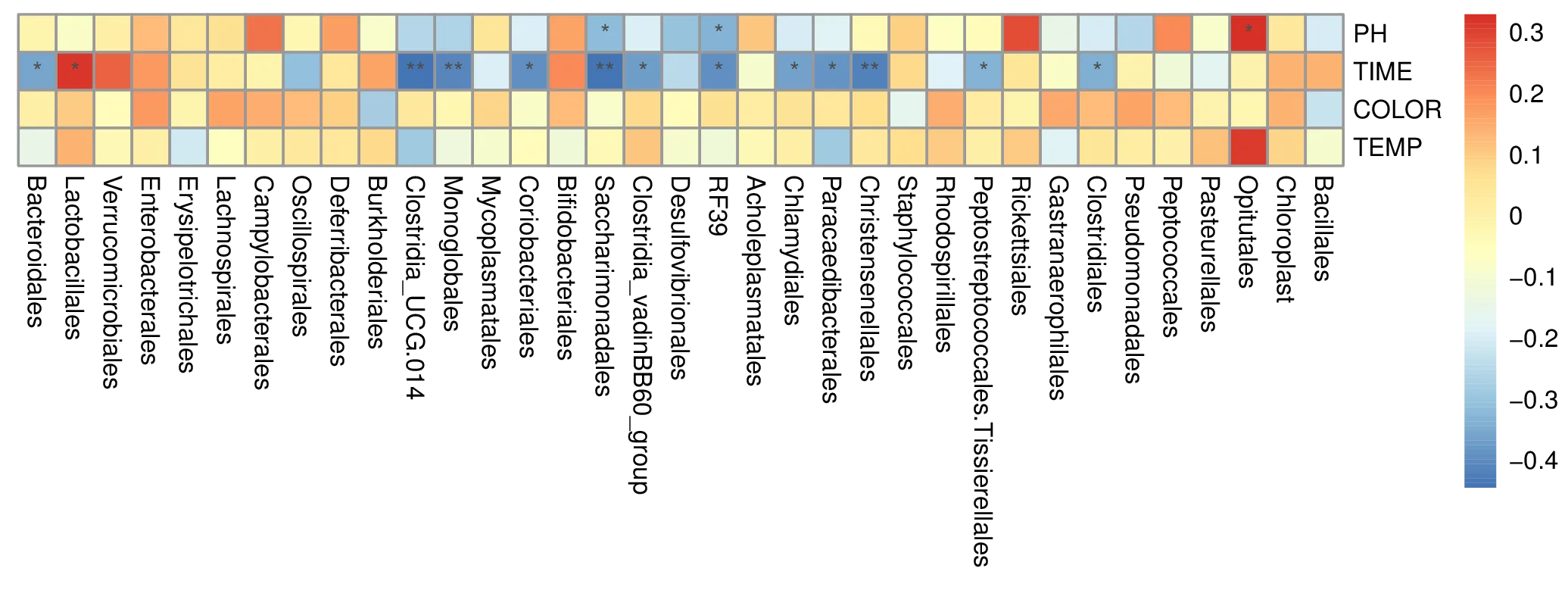
アンプリコンメタゲノムシーケンスは、OTUまたはASVを使用して微生物種の同定および分化を目指す研究者にとって強力なツールです。16S/18S/ITS rRNAシーケンス結果を用いて、アルファ(α)およびベータ(β)多様性解析による環境の微生物多様性を特徴付け、目的の微生物を同定、記述、追跡することができます。Novogene Amplicon Metagenomic Sequencingサービスを使用した主な論文のリストです。
-
Biosynthesis and recycling of magnetite nanocatalysts from Fe-rich sludge
Resources, Conservation, and RecyclingIssue Date: 14 April 2022IF: 13.716DOI: 10.1016/j.resconrec.2022.106348
-
Journal of Hazardous MaterialsIssue Date: 3 February 2022IF: 10.58DOI: 10.1016/j.jhazmat.2022.128396
-
Pharmaceuticals (Basel)Issue Date: 11 July 2022IF: 5.215DOI: 10.3390/ph15070850
-
Association of female reproductive tract microbiota with egg production in layer chickens
GigaScienceIssue Date: 23 September 2021IF: 7.658DOI: 10.1093/gigascience/giab067
-
Frontiers in ImmunologyIssue Date: 8 March 2021IF: 7.97DOI: 10.3389/fimmu.2021.640767
-
Cell Host & MicrobeIssue Date: 10 April 2019IF: 15.923DOI: 10.1016/j.chom.2019.02.003
-
Bioresource TechnologyIssue Date: January 2019IF: 7.539DOI: 10.1016/j.biortech.2018.10.045
-
MicrobiomeIssue Date: 03 April 2018IF: 11.607DOI: 10.1186/s40168-018-0441-4
-
Nature CommunicationsIssue Date: 09 January 2017IF: 12.121DOI: 10.1038/ncomms13839
-
Gut microbiota dysbiosis contributes to the development of hypertension
MicrobiomeIssue Date: 01 February 2017IF: 11.607DOI: 10.1186/s40168-016-0222-x
Novogene's Qiime2 Analysis Pipeline
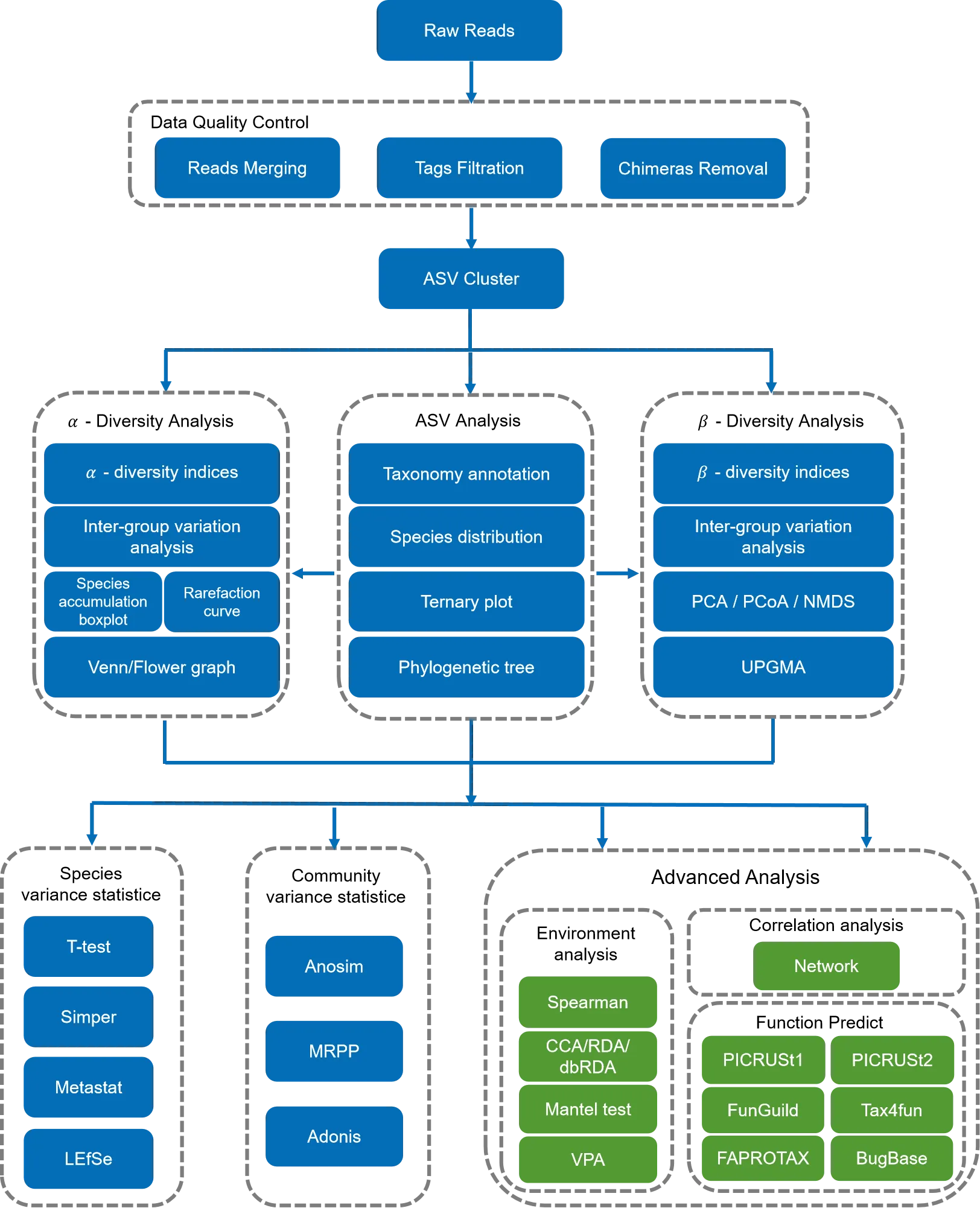
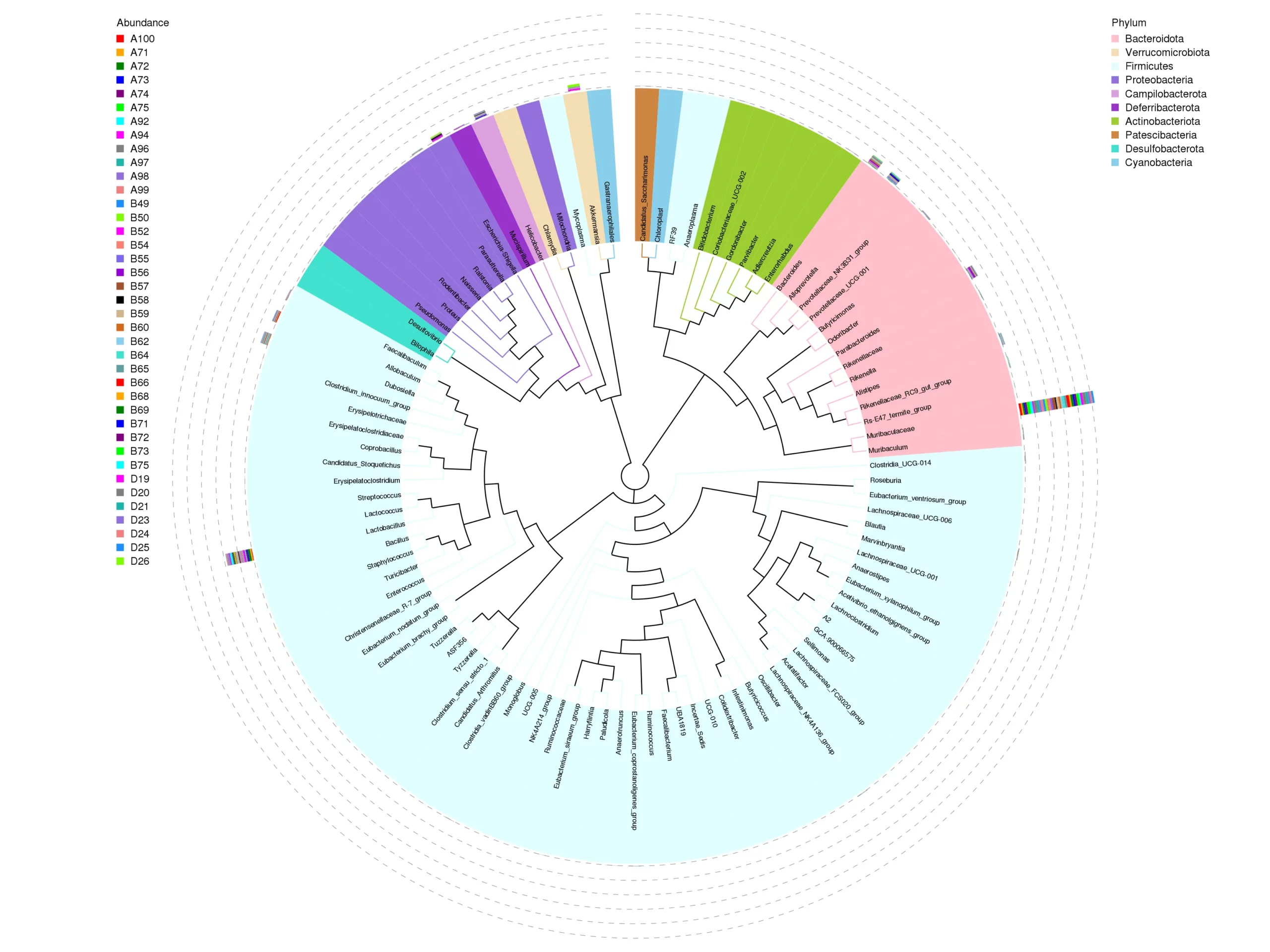
The evolutionary tree in genus
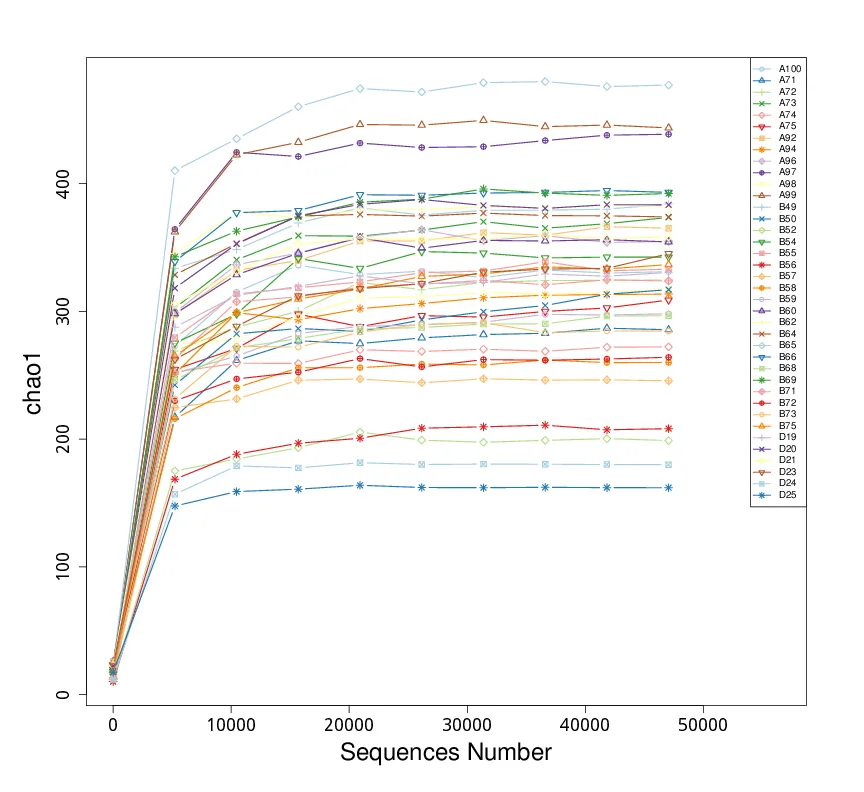
Alpha Diversity- Rarefaction Curves
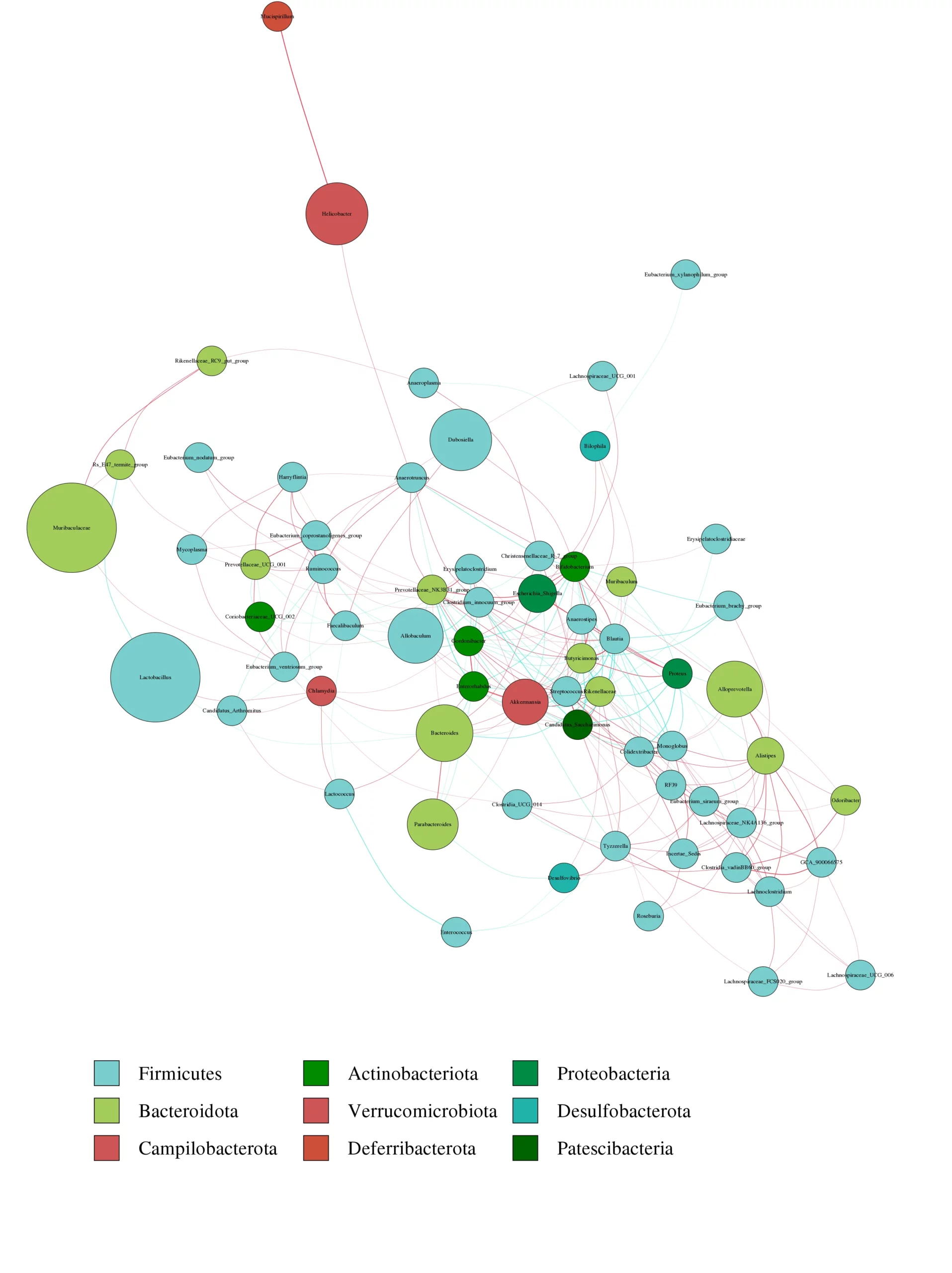
Network Analysis
Spearman Correlation Test